Abstrak
Penyakit karat mahkota, disebabkan oleh Puccinia coronata f. sp. avenae , menimbulkan ancaman signifikan terhadap produksi oat global ( Avena sativa L.). Penanda molekuler sangat penting untuk membantu integrasi beberapa gen resistensi ke dalam satu kultivar oat untuk mencapai ketahanan resistensi genetik. Di sini, kami memvalidasi penanda yang dilaporkan sebelumnya untuk gen resistensi spesifik ras Pc39 , Pc45 / PcKM , Pc54 , dan Pc68 dan mengembangkan penanda PCR spesifik alel kompetitif baru yang terkait erat dengan lokus ini. Penanda ini kemudian disaring di seluruh koleksi 150 kultivar oat. Analisis data penanda molekuler, informasi silsilah, dan profil resistensi penyakit mengidentifikasi beberapa kultivar oat yang kemungkinan membawa Pc39 dan Pc68 . Pembawa Pc68 yang baru diidentifikasi mencakup kultivar Galileo dan Graza80, sementara Warrego, Kowari, Possum, Forester, Drummond, Hokonui, Glider, dan Culgoa diidentifikasi sebagai pembawa potensial Pc39 . Perbedaan antara postulasi sebelumnya tentang pembawa gen Pc dan analisis fenotipe dan genotipe kami juga ditemukan. Misalnya, galur gandum Glider sebelumnya dipostulatkan membawa Pc58 atau Pc59 , tetapi positif untuk penanda terkait Pc39 dan memiliki profil resistensi yang sama dengan pembawa Pc39 . Temuan ini menggarisbawahi pentingnya pemanfaatan penanda molekuler dalam melacak gen resistensi dalam plasma nutfah pemuliaan.
Ringkasan Bahasa Sederhana
Oat merupakan tanaman serealia penting yang produksinya menurun akibat kerugian yang disebabkan oleh penyakit karat mahkota. Penyakit ini disebabkan oleh jamur karat bernama Puccinia coronata f. sp. avenae . Untuk mengembangkan kultivar oat yang tahan terhadap karat mahkota oat, program pemuliaan sangat bergantung pada penggunaan penanda DNA yang terkait dengan ketahanan genetik, yang penting untuk memilih tanaman yang membawa gen ketahanan yang diinginkan. Mengingat pengetahuan tentang penanda DNA untuk gen ketahanan karat mahkota oat ( Pc ) terbatas, penelitian ini memvalidasi dan mengembangkan penanda baru untuk beberapa gen Pc ini . Lebih jauh, kumpulan 150 kultivar oat disaring menggunakan penanda DNA ini untuk mengidentifikasi pembawa gen Pc target .
Singkatan
Krom
kromosom
Universitas Gadjah Mada
genomik komparatif
DArTSeq
teknologi array keragaman sekuensing
Bahasa Inggris GBS
genotipe dengan sekuensing
GWAS
studi asosiasi genom secara luas
KASP
PCR spesifik alel kompetitif
MAS
pemilihan berbantuan penanda
Pca
Puccinia coronata f. sp. jalan
KOLAM
Silsilah Garis Gandum
QTL
lokus sifat kuantitatif
SNP
polimorfisme nukleotida tunggal
STS
situs yang diberi tag urutan
RUMBAI
Analisis Sifat Berdasarkan Asosiasi, Evolusi, dan Keterkaitan
1. PENDAHULUAN
Penyakit jamur yang disebabkan oleh Puccinia coronata f. sp. avenae ( Pca ), yang dikenal sebagai karat mahkota, adalah salah satu ancaman global utama bagi produksi gandum ( Avena sativa L.) (Nazareno et al., 2018 ; Simons, 1985 ). Tingkat keparahan infeksi karat mahkota gandum dipengaruhi oleh kondisi cuaca, beban inokulum, dan genotipe kultivar gandum yang ditanam. Epidemi penyakit karat mahkota biasanya dapat mengurangi hasil gandum sebesar 10%–50%; namun, seluruh ladang dapat hilang dalam kondisi lingkungan yang optimal untuk infeksi tanaman (Chambers & Thomas, 2020 ; Strunk et al., 2022 ). Menanam kultivar gandum dengan ketahanan genetik dianggap sebagai metode yang paling ramah lingkungan dan hemat biaya untuk pengendalian karat (Figueroa et al., 2020 ; Nazareno et al., 2018 ).
Resistensi spesifik ras yang diberikan oleh gen tunggal-mayor mengikuti model interaksi gen-untuk-gen (Flor, 1971 ; Dodds, 2023 ), dan telah menjadi strategi umum untuk mengendalikan karat mahkota di lapangan (Nazareno et al., 2018 ). Namun, jenis resistensi genetik ini tidak memiliki daya tahan jangka panjang karena populasi karat dapat dengan cepat mengembangkan varian gen efektor avirulensi baru dan menghindari deteksi tanaman (Figueroa et al., 2020 ). Evolusi fenotipe virulensi baru dalam populasi Pca dan kekalahan resistensi telah didokumentasikan dengan baik di banyak bagian dunia (misalnya, Henningsen et al., 2024 ; Klenová-Jiráková et al., 2010 ; Moreau et al., 2024 ; Nguyen et al., 2025 ; van Niekerk et al., 2001 ) dan merupakan tantangan saat pemuliaan untuk perlindungan tanaman genetik. Sebaliknya, resistensi non-spesifik ras, juga dikenal sebagai resistensi tanaman dewasa atau parsial, umumnya melibatkan resistensi parsial spektrum luas yang dapat mengurangi inokulum karat dan oleh karena itu tekanan penyakit di lapangan (Nazareno et al., 2022 ). Dengan demikian, pendekatan yang tepat untuk memperpanjang daya tahan gen spesifik ras adalah penumpukan kedua jenis sumber resistensi menjadi satu kultivar (Mundt, 2018 ; Periyannan et al., 2017 ). Seleksi berbantuan penanda (MAS) merupakan metode yang efektif dan berthroughput tinggi untuk memperlancar proses ini (misalnya, Gnanesh et al., 2013 ). Meskipun demikian, penerapan MAS dalam pemuliaan gandum untuk ketahanan terhadap karat mahkota masih terbatas karena kurangnya penanda berkualitas tinggi dan berthroughput tinggi.
Lebih dari 90 gen ketahanan tanaman terhadap penyakit karat mahkota telah dikatalogkan dalam gandum (Carson, 2017 ). Baru-baru ini, Wight dkk. ( 2024 ) menyusun inventaris terbaru gen dan lokus sifat kuantitatif (QTL), termasuk gen untuk penyakit karat mahkota, dalam gandum heksaploid, yang merinci posisi kemungkinan gen tersebut pada peta konsensus 2018 (Bekele dkk., 2018 ) dan genom OT3098 v2 pada situs web basis data GrainGenes ( https://wheat.pw.usda.gov/jb/?data=/ggds/oat-ot3098v2-pepsico ) (Yao dkk., 2022 ). Dari jumlah tersebut, sekumpulan kecil gen (termasuk Pc39 , Pc45 , Pc54 , Pc68 , dan lainnya) telah dipetakan ke lokasi kromosom (chr) dalam persilangan biparental atau dengan pemetaan asosiasi (Admassu-Yimer et al., 2018, 2022 ; Bush & Wise, 1998 ; Chen et al., 2006 ; Gnanesh et al., 2013, 2015 ; Hoffman et al., 2006 ; Kebede et al., 2019 ; Klos et al., 2017 ; Kulchesiki et al., 2010 ; McCartney et al., 2011 ; McNish et al., 2020 ; Satheeskumar et al., 2011 ; Sowa & Paczos-Grzeda, 2020 ; Wight dkk., 2024 ; Zhao dkk., 2020 ). Akan tetapi, hanya beberapa polimorfisme nukleotida tunggal (SNP) yang terkait dengan gen Pc ini yang telah diubah menjadi penanda yang dapat segera diaplikasikan untuk MAS. Sistem genotipe PCR spesifik alel kompetitif (KASP) untuk SNP merupakan salah satu teknologi yang paling banyak diadopsi yang dapat mendukung MAS dalam pemuliaan gandum (Rahman dkk., 2023 ).
Penanda SNP untuk Pc39 dipetakan ke Mrg11 (Bekele et al., 2018 ; Chaffin et al., 2016 ; Sowa & Paczos-Grzeda, 2020 ; Zhao et al., 2020 ) dan beberapa penanda SNP terkait Pc39 diubah menjadi penanda KASP dan divalidasi dalam panel yang terdiri dari 74 galur dan kultivar oat (Zhao et al., 2020 ). Di antara ini, penanda GMI_ES01_c12570_390 dan GMI_ES15_c6153_3929 diusulkan menjadi yang paling berguna untuk MAS. Baru-baru ini, Wight et al. ( 2024 ) menggunakan pemetaan genetik dan fisik komparatif pada genom referensi oat OT3098 v2 (PepsiCo, https://wheat.pw.usda.gov/jb?data=/ggds/oat-ot3098v2-pepsico ), yang disimpan dalam basis data GrainGenes (Yao et al., 2022 ), untuk menetapkan penanda KASP ini dan beberapa penanda daerah teramplifikasi berkarakter sekuens yang diasosiasikan dengan Pc39 (Sowa & Paczos-Grzęda, 2020 ) ke daerah genom homolog pada chr4C, chr6A, dan chr5D. Dalam studi lain, Kebede et al. ( 2019 ) memetakan gen Pc45 dan mengonfirmasi identitasnya dengan PcKM (Gnanesh et al., 2015 ). Dua penanda KASP I05-0874-KOM16c1 dan GMI_ES02_c16987_268 pada Mrg08 (chr2D, OT3098 v2) dilaporkan sebagai penanda yang paling spesifik untuk keberadaan/ketiadaan Pc45 / PcKM dalam kumpulan 71 galur gandum (Gnanesh et al., 2015 ; Kebede et al., 2019 ). Gen Pc54 dipetakan ke Mrg02 (chr7D, OT3098 v2) oleh Admassu-Yimer et al. ( 2022 ), dengan tiga array-SNP ( GMI_ES03_c95_413 , GMI_ES15_c15279_258 , dan GMI_ES22_c2813_554 ) dilaporkan terkait erat dengan Pc54 (Admassu-Yimer et al., 2022 ). Dalam studi asosiasi genom-lebar (GWAS), Klos et al. ( 2017 ) mengidentifikasi SNP yang berasal dari genotyping by sequencing (GBS), Avgbs_228849 , pada Mrg19 (chr3D, OT3098 v2) yang secara signifikan terkait dengan resistensi Pc68 terhadap beberapa isolat Pca Amerika Utara .
Dalam studi ini, kami mengonversi SNP yang dikaitkan dengan Pc54 dan Pc68 menjadi penanda KASP dan menggunakan penanda ini dan penanda KASP yang tersedia untuk Pc39 dan Pc45 untuk membuat genotipe sekumpulan 86 galur diferensial karat mahkota gandum. Ini dikombinasikan dengan data genotipe genome-wide diversity arrays technology sequencing (DArTSeq) dari galur-galur ini (Nguyen et al., 2024 ) untuk analisis pemetaan asosiasi. Analisis ini memvalidasi lokasi genom yang diidentifikasi sebelumnya untuk Pc39 , Pc45 , Pc54 , dan Pc68 dan memungkinkan pengembangan penanda KASP baru yang spesifik untuk gen-gen Pc ini . Koleksi beragam dari 150 galur gandum (Nguyen et al., 2024 ) disaring menggunakan penanda yang paling spesifik untuk mengidentifikasi sumber potensial resistensi genetik. Informasi silsilah dan profil resistensi penyakit galur gandum kemudian dibandingkan untuk lebih mendukung dugaan gen Pc yang ada dalam galur gandum ini. Secara keseluruhan, temuan dari studi ini menyediakan sumber daya tambahan untuk pemuliaan ketahanan terhadap karat mahkota gandum dengan bantuan penanda.
2 BAHAN DAN METODE
2.1 Tumbuhan dan materi DNA
Dua set diferensial oat dari Australia dan Amerika Serikat yang terdiri dari 86 galur telah dideskripsikan sebelumnya (Nguyen et al., 2024 ). Koleksi oat dengan galur yang bersumber dari Departemen Pertanian AS—Layanan Penelitian Pertanian, Bank Gen Gandum Australia, dan stok benih “ Avena ” di Organisasi Penelitian Ilmiah dan Industri Persemakmuran (Tabel S1 ) baru-baru ini disusun oleh Nguyen et al. ( 2024 ). DNA diekstraksi dari bibit berumur 3 minggu seperti yang dideskripsikan oleh Ellis et al. ( 2005 ) dan kualitas serta konsentrasi dinilai menggunakan Spektrofotometer NanoDrop 8000 (NanoDrop Technologies Inc.) dan uji fluoresensi PicoGreen (Singer et al., 1997 ).

2.2 Uji KASP untuk Pc39 , Pc45 / PcKM , Pc54 , dan Pc68
Kami mengadopsi penanda KASP-ready untuk gen Pc39 , yang dikenal sebagai GMI_ES01_c12570_390 dan GMI_ES15_c6153_392 yang dikembangkan oleh Zhao et al. ( 2020 ), dan untuk Pc45/PcKM , GMI_ES02_c16987_268 dan I05-0874-KOM16c1 yang dikembangkan oleh Gnanesh et al. ( 2015 ) dan Kebede et al. ( 2019 ), masing-masing. Selain itu, kami merancang penanda KASP baru untuk SNP GMI_ES15_c15279_258 , GMI_ES03_c95_413 , GMI_ES22_c2813_554 , yang terkait erat dengan Pc54 (Admassu-Yimer et al., 2022 ), dan Avgbs_228849 , yang dilaporkan berasosiasi secara signifikan dengan Pc68 (Klos et al., 2017 ). Urutan flanking dari SNP ini (diekstraksi dari data array SNP dari Tinker et al. [ 2014 ] dan basis data T3/Oat [Morales et al., 2022 ]) diimpor ke dalam sistem perangkat lunak Kraken untuk desain penanda menggunakan parameter default (LGC Biosearch Technologies; https://www.biosearchtech.com/ ). Penanda KASP tambahan KASP_chr6A_443082340 ( Pc39 ), KASP_chr6A_443082349 ( Pc39 ), KASP_chr6A_443117388 ( Pc39 ), KASP_chr2D_181078165 ( Pc45 ), KASP_chr7D_464499341 ( Pc54 ), KASP_chr3D_466310997 ( Pc68 ), dan KASP_chr3D_473653096 ( Pc68 ) (Tabel S2 ) dirancang berdasarkan SNP turunan DArTSeq yang secara signifikan terkait dengan gen yang diinginkan. Pengujian KASP dilakukan dalam Sistem Genotipe PCR SNPline (LGC), mengikuti metode yang dijelaskan oleh Shi et al. ( Tahun 2023 ).
2.3 Analisis pemetaan asosiasi
Asosiasi genotipe dengan resistensi Pc dinilai dalam 86 galur diferensial oat menggunakan data genotipe DArTSeq genomik yang dihasilkan sebelumnya untuk galur-galur ini (Nguyen et al., 2024 ) bersama dengan genotipe untuk delapan penanda KASP yang berasal dari SNP yang terkait dengan Pc39 (Zhao et al., 2020 ), Pc45 (Gnanesh et al., 2015 ; Kebede et al., 2019 ), Pc54 (Admassu-Yimer et al., 2022 ), dan Pc68 (Klos et al., 2017 ). Setiap garis oat diberi skor fenotip (1 atau 2) berdasarkan deskripsi sebelumnya sebagai pembawa (skor = 2) atau bukan pembawa (skor = 1) gen Pc39 , Pc45 , Pc54 , atau Pc68 (Carson, 2017 ; Chen et al., 2006 ; Kebede et al., 2019 ; Mitchell Fetch et al., 2007 ; Park, 2013 ). Analisis asosiasi dilakukan dengan menggunakan perangkat lunak Trait Analysis by Association, Evolution, and Linkage (TASSEL) (v5.2.63) (Bradbury et al., 2007 ). Dalam TASSEL, struktur populasi dan kekerabatan dihitung dari data penanda yang diimpor menggunakan empat komponen utama dan pendekatan identitas terpusat menurut keadaan, masing-masing, sebagai masukan untuk alur kerja model linier campuran. Tidak ada kompresi yang dilakukan, dan komponen varians diestimasi ulang setelah setiap penanda. Ambang koreksi Bonferroni dihitung dengan membagi nilai p ( α = 0,01) dengan jumlah penanda yang digunakan dalam analisis asosiasi. Untuk setiap gen Pc , analisis asosiasi dibatasi pada penanda SNP yang terletak pada kromosom tempat gen tersebut sebelumnya dipetakan (misalnya, chr2D untuk Pc45 ).
2.4 Investigasi silsilah
Hubungan antara kultivar oat divisualisasikan menggunakan perangkat lunak tampilan silsilah Helium (Shaw et al., 2014 ). Catatan silsilah diperoleh dari Fitzsimmons et al. ( 1983 ) dan basis data T3/Oat (Morales et al., 2022 ), dengan informasi yang diekstrak dari basis data “Pedigree of Oat Lines” (POOL) ( https://triticeaetoolbox.org/POOL/index_db.php ; Tinker & Deyl, 2005 ). Silsilah untuk setiap aksesi dipecahkan ke Induk 1 dan Induk 2 untuk membuatnya kompatibel dengan Helium, dan basis data yang dihasilkan kemudian diimpor ke Helium. Bersama dengan silsilah, postulat gen resistensi sebelumnya diimpor ke Helium untuk pemeriksaan dalam konteks silsilah.
2.5 Uji infeksi karat
Satu set 16 isolat Pca yang dikumpulkan dari daerah penghasil gandum utama di Australia pada tahun 2022 dan 2023 digunakan (Henningsen et al., 2024 ; Nguyen et al., 2025 ). Metode infeksi dan penilaian sebelumnya dijelaskan oleh Miller et al. ( 2020 ). Skor tersebut kemudian diubah menjadi skala numerik 0–9 seperti yang dijelaskan oleh Miller et al. ( 2018 ) dan diplot sebagai peta panas menggunakan ComplexHeatmap (v2.14.0) di R (4.2.2) (Gu, 2022 ).
2.6 Analisis perbandingan sekuens
Lokasi fisik penanda yang terkait dengan gen Pc yang diinginkan ditentukan oleh penelusuran BLAST (basic local alignment search tool) (Altschul et al., 1990 ) dari urutan flanking SNP terhadap genom referensi OT3098 v2 dari basis data GrainGenes (Yao et al., 2022 ), menggunakan Geneious Prime 2022.2.2. Urutan penanda flanking diperoleh dari NCBI GenBank (di mana NCBI adalah Pusat Informasi Bioteknologi Nasional) ( https://www.ncbi.nlm.nih.gov/nuccore/ ) dan basis data T3/Oat (Morales et al., 2022 ). Hubungan sintenik ditetapkan melalui platform genomik komparatif (CoGe) ( https://genomevolution.org ). Genom referensi OT3098 v2 diimpor ke CoGe, dan alat SynMap2 (Haug-Baltzell et al., 2017 ) digunakan untuk penyelarasan kromosom dan visualisasi sintenik https://genomevolution.org/CoGe/SynMap.pl .
3 HASIL
3.1 Identifikasi penanda baru untuk karat mahkota gandum melalui analisis pemetaan asosiasi
Untuk memvalidasi penanda yang dilaporkan sebelumnya dan mengidentifikasi penanda baru yang lebih erat kaitannya dengan Pc39 , Pc45 , Pc54 , dan Pc68 , kami melakukan analisis asosiasi pada 86 galur dari set diferensial penyakit karat mahkota gandum yang masing-masing membawa gen Pc yang telah ditentukan . Kami pertama-tama menilai genotipe set ini menggunakan penanda siap KASP untuk Pc39 , GMI_ES01_c12570_390 , dan GMI_ES15_c6153_392 (Zhao et al., 2020 ), dan Pc45 , GMI_ES02_c16987_268 , dan I05-0874-KOM16c1 (Gnanesh et al., 2015 ; Informasi Pendukung S1 ). Kami selanjutnya merancang penanda KASP baru untuk Pc54 dan Pc68 dari SNP yang terkait erat GMI_ES15_c15279_258 , GMI_ES03_c95_413 , GMI_ES22_c2813_554 (Admassu-Yimer et al., 2022 ), dan avgbs_228849 (Klos et al., 2017 ), yang berhasil mendeteksi genotipe SNP ini di seluruh set diferensial. Genotipe penanda ini digabungkan dengan genotipe SNP DArTSeq di seluruh genom yang sebelumnya dibuat untuk galur ini (Nguyen et al., 2024 ) untuk melakukan analisis asosiasi. Karena lokasi kromosom gen-gen ini telah diketahui, analisis asosiasi untuk setiap gen dibatasi pada penanda pada kromosom relevan tempat gen-gen tersebut sebelumnya dipetakan (Gambar 1 ; Gambar S1 ).
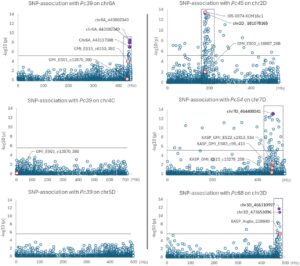
Dalam laporan baru-baru ini, Wight et al. ( 2024 ) menetapkan posisi fisik beberapa urutan penanda yang terkait dengan lokus Pc39 (Sowa & Paczos-Grzeda, 2020 ; Zhao et al., 2020 ) ke daerah homoeolog chr4C, chr5D, dan chr6A dari genom referensi OT3098 v2. Perbandingan urutan kromosom ini mengonfirmasi bahwa masing-masing mengandung daerah yang relatif kecil yang sintenus di seluruh ketiga kromosom dan mencakup lokasi urutan penanda yang diketahui (Gambar S2a ). Oleh karena itu, kami melakukan analisis asosiasi untuk Pc39 menggunakan penanda DArTSeq pada chr4C, chr5D, dan chr6A, bersama dengan penanda KASP untuk lokus ini. Dalam analisis ini, asosiasi signifikan SNP DArTSeq dengan Pc39 hanya terjadi pada chr6A di wilayah antara 436–443 Mb, tanpa asosiasi signifikan yang terdeteksi di wilayah homolog pada chr5D dan chr4C (Gambar 1 ). Penanda GMI_ES01_c12570_390 dan GMI_ES15_c6153_392 (Zhao et al., 2020 ) keduanya terjadi di wilayah chr6A ini; namun, penanda SNP DArTSeq lain dari wilayah ini (Informasi Pendukung S2 ) menunjukkan asosiasi tinggi dengan Pc39 dalam set diferensial.
SNP DArTSeq yang terkait dengan Pc68 terletak di wilayah 463–478 Mb dari chr3D (Gambar 1 ). Penanda KASP_avgbs_228849 untuk Pc68 dikembangkan dari SNP GBS (Klos et al., 2017 ) dan SNP yang sama juga ditemukan sebagai penanda DArTSeq pada 470.133.974 bp pada chr3D dalam penelitian ini. Panggilan genotipe untuk penanda DArTSeq ini dan KASP_avgbs_228849 identik, kecuali untuk beberapa panggilan yang hilang dalam uji KASP. Beberapa penanda SNP DArTSeq menunjukkan asosiasi yang lebih tinggi dengan Pc68 daripada KASP_avgbs_228849 .
Penanda DArTSeq yang berasosiasi secara signifikan untuk Pc45 dan Pc54 ditemukan antara 180–200 Mb pada chr2D dan 435–495 Mb pada chr7D, masing-masing (Gambar 1 ). Wilayah ini juga terkolokasi dengan penanda KASP yang sesuai dengan Pc45 pada chr2D dan Pc54 pada chr7D. Meskipun beberapa penanda di luar wilayah target melewati ambang batas signifikansi, tanpa penyelidikan lebih lanjut, sulit untuk mengatakan apakah mereka mewakili asosiasi sejati dalam ketidakseimbangan hubungan jarak jauh dengan gen penyebab atau mewakili positif palsu yang dipengaruhi oleh struktur populasi. Untuk Pc45 , penanda I05-0874-KOM16c1 yang ada menunjukkan skor signifikansi tertinggi dibandingkan dengan SNP DArTSeq dalam analisis asosiasi. Namun, untuk Pc54 , beberapa penanda DArTSeq (Informasi Pendukung S2 ) menunjukkan asosiasi yang lebih tinggi daripada penanda lain yang dijelaskan sebelumnya.
3.2 Validasi penanda KASP menggunakan set diferensial oat
Dari analisis asosiasi, beberapa SNP DArTSeq yang paling kuat berasosiasi dengan gen Pc dipilih dan diubah menjadi penanda KASP (Gambar 1 ; Tabel S2 ). Penanda KASP yang baru dikembangkan ini digenotipe terhadap set diferensial dan dibandingkan dengan hasil penanda yang dijelaskan sebelumnya (Gambar S3 ).
3.2.1 Pc 39
Uji KASP dari penanda GMI_ES01_c12570_390 menampilkan tiga genotipe berbeda ( AA / AG / GG ) di antara galur diferensial (Gambar S3a ; Informasi Pendukung S1 ). Kehadiran genotipe yang tampaknya heterozigot ( AG ) dalam galur diferensial Pc39 dan 33 galur lainnya mungkin disebabkan oleh amplifikasi sekuens serupa pada chr4C dan chr6A yang berbeda oleh SNP ini dalam galur oat individual, seperti halnya dalam referensi OT3098 v2 (Gambar S2b ). Oleh karena itu, penanda GMI_ES01_c12570_390 dianggap tidak efektif untuk diskriminasi Pc39 dalam uji KASP.
Di sisi lain, penanda KASP GMI_ES15_c6153_392 lebih spesifik daripada GMI_ES01_c12570_390 , mendeteksi 17 garis diferensial positif dibandingkan dengan 52 (Tabel 1 ; Informasi Pendukung S1 ). Dari garis positif GMI_ES15_c6153_392 , dua adalah garis diferensial Pc39 yang ditentukan dari set AS dan AUS, dan dua lainnya, Cleanleaf dan Leggett, dipostulatkan membawa Pc39 berdasarkan fenotipe (Mitchell Fetch et al., 2007 ; Park, 2013 ). Namun, penanda ini tidak positif di Barcoo, yang sebelumnya dipostulatkan membawa Pc39 , Pc61 , dan PcBett (Park, 2013 ). Garis positif lainnya membawa gen resistensi yang berbeda (misalnya, Pc45 , Pc58 , Pc60 , Pc61 , Pc70 , Pc91 , Pc96 ) atau gen yang tidak diketahui, seperti Culgoa ( PcCulgoa ), Genie, dan Warrego (keduanya dianggap memiliki Pc61 + gen yang tidak diketahui; Park et al., 2009 ) (Informasi Pendukung S1 ).
| gen pc | Referensi | Penanda | Lokasi fisik (OT3098 V2) | Jumlah garis positif untuk alel resistensi dalam set diferensial ( n = 86) |
|---|---|---|---|---|
| Pc39 | Zhao dan kawan-kawan ( 2020 ) | GMI_ES01_c12570_390 | chr6A: 433390138–433390238/chr4C:1945282 –1945382 | 52 sebuah |
| GMI_ES15_c6153_392 | bab 6A: 436047504–436047387 | 17 | ||
| Pc45 / PcKm | Kebede dan kawan-kawan ( 2019 ) | GMI_ES02_c16987_268 | nomor 2D: 179682216–179682335 | 43 |
| I05-0874-KOM16c1 | nomor 2D: 181118507–181118369 | 3 | ||
| Studi ini | KASP_chr2D_181078165 | nomor 2D: 181078104–181078224 | 3 | |
| Pc54 | Admassu-Yimer et al. ( 2022 ) dan penelitian ini | KASP_GMI_ES15_c15279_258 | chr7D: 461451176–461450972/chr7D: 489346440–489346236 | 44 sebuah |
| KASP_GMI_ES03_c95_413 | chr7D: 462940037–462939953/chr7D: 490417282–490417198 | 24 | ||
| KASP_GMI_ES22_c2813_554 | chr7D: 465112841–465112723/chr7D: 494022361–494022243 | 12 | ||
| Studi ini | KASP_chr7D_464499341 | chr7D: 464499281–464499400/chr7D: 493116025–493116144 | 2 | |
| Pc68 | Klos dan kawan-kawan ( 2017 ) | KASP_Rata-rata_228849 | nomor 3D: 470133926–470134034 | 8 |
| Studi ini | KASP_chr3D_466310997 | kronologi3D: 466310942–466311050 | 3 | |
| KASP_chr3D_473653096 | nomor 3D: 473653029–473653163 | 3 |
Catatan : “/” memisahkan lokasi dengan keyakinan tertinggi yang sama dari pencarian BLAST (alat pencarian penyelarasan lokal dasar) dalam genom referensi OT3098 V2. a Kehadiran heterozigositas pada beberapa garis yang dapat disebabkan oleh duplikasi urutan penanda pada homoeo-alel atau secara lokal.
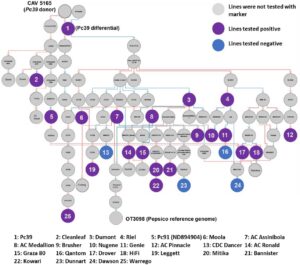
Pencarian pada database “POOL” ( https://triticeaetoolbox.org/POOL/index_db.php ), mengungkapkan hubungan silsilah antara Genie, Warrego, HiFi, dan Pc91 (ND894904) dengan operator Pc39 lainnya (Gambar 2 ).
Tiga SNP DArTSeq ( chr6A_443082340 , chr6A_443082349 , chr6A_443117388 ) menunjukkan asosiasi yang jauh lebih tinggi dengan Pc39 daripada penanda KASP yang ada. Namun, penanda KASP baru yang dihasilkan dari SNP ini hanya mendeteksi genotipe SNP yang terkait dengan resistensi di semua galur yang diuji (data tidak ditampilkan), yang tidak konsisten dengan data genotipe DArTSeq.
3.2.2 Pc 45
Perbandingan hasil genotipe untuk KASP_chr2D_181078165 (dikembangkan dari SNP DArTSeq dalam penelitian ini) dengan penanda I05-0874-KOM16c1 dan GMI_ES02_c16987_268 (Gnanesh et al., 2015 ; Kebede et al., 2019 ) pada set diferensial menunjukkan bahwa KASP_chr2D_181078165 dan I05-0874-KOM16c1 sama-sama spesifik untuk Pc45 / PcKM (Tabel 1 ; Informasi Pendukung S1 ), karena keduanya hanya positif untuk dua garis diferensial Pc45 (USA dan AUS), ditambah satu garis oat tambahan, IA B605X, yang merupakan donor PcKM (Gnanesh et al., 2015 ; McMullen et al., 2005). ). Sebaliknya, GMI_ES02_c16987_268 tidak spesifik untuk Pc45 / PcKM , karena alel resistensi juga muncul pada 34 galur oat lainnya, yang dianggap memiliki sumber resistensi lainnya (Informasi Pendukung S1 ).
3.2.3 Pc 54
Penanda baru KASP_chr7D_464499341 menunjukkan spesifisitas yang lebih tinggi untuk Pc54 daripada penanda yang diterbitkan sebelumnya GMI_ES03_c95_413 , GMI_ES15_c15279_258 , dan GMI_ES22_c2813_554 (Admassu-Yimer et al., 2022 ) (Tabel 1 ; Gambar S3c ) karena hanya positif untuk dua garis diferensial Pc 54 dalam set ini. Sebaliknya, penanda KASP_GMI_ES22_c2813_554 positif untuk 12 galur (Tabel 1 ), termasuk galur diferensial Pc54 dan 10 galur lainnya (Informasi Pendukung S1 ), sementara alel resistensi KASP_GMI_ES03_c95_413 dan KASP_GMI_ES15_c15279_258 masing-masing terdeteksi pada 24 dan 44 galur oat, selain galur diferensial Pc54. Semua sekuens penanda Pc54 ditemukan di dua lokasi identik di dekat ujung distal chr7D, berjarak sekitar 28 Mbp (Tabel S1 ). Penjajaran kromosom menunjukkan duplikasi tandem besar di wilayah chr7D ini (Gambar S2a ). Selain itu, penanda KASP_GMI_ES15_c15279_258 menunjukkan heterozigositas di beberapa galur (Gambar 3c ). Hal ini kemungkinan merupakan konsekuensi dari polimorfisme antara daerah duplikat pada chr7D, seperti halnya pada genom referensi OT3098 (Gambar S2b ).
3.2.4 Pc 68
Dua penanda Pc68 baru , KASP_chr3D_473653096 dan KASP_chr3D_466310997 , sangat spesifik untuk pembawa Pc68 yang diketahui , hanya mendeteksi dua garis diferensial Pc68 dan garis oat yang Leggett postulatkan membawa Pc39 , Pc68 , dan Pc94 (Mitchell Fetch et al., 2007 ). Sebaliknya, penanda yang dipublikasikan KASP_avgbs_228849 (Klos et al., 2017 ) positif untuk ketiga garis ini serta lima garis tambahan: Amagalon ( Pc91 ), Bondvic, H548 ( Pc Tidak Diketahui ), Trispernia ( Pc6D ), dan H441 ( Pc53 ), tidak satupun dari yang telah dipostulatkan membawa Pc68 . Amagalon, heksaploid sintetis dari Avena magna / A vena longiglumis , adalah donor gen Pc91 (Rooney et al., 1994 ), dan Bondivic (Anthony/Bond//Boone) adalah garis diferensial awal untuk karat mahkota gandum yang digunakan oleh Simons & Murphy ( 1955 ); namun, data silsilah yang tersedia untuk publik yang terbatas pada garis lainnya membuat sulit untuk menentukan hubungan apa pun dengan pembawa Pc68 ( https://triticeaetoolbox.org/POOL/index_db.php ) (Tabel 1 ; Informasi Pendukung S1 ).
3.3 Penerapan penanda KASP diagnostik untuk gen ketahanan karat mahkota gandum dalam skrining koleksi gandum
Setelah validasi penanda KASP dalam set diferensial, penanda paling spesifik untuk Pc39 ( GMI_ES15_c6153_392 ), Pc45 ( I05-0874-KOM16c1 dan KASP_2D_181078165 ), Pc54 ( KASP_chr7D_464499341 ), dan Pc68 ( KASP_chr3D_466310997 dan KASP_chr3D_473653096 ) digunakan untuk menyaring koleksi yang lebih luas dari 150 kultivar gandum untuk mengidentifikasi pembawa potensial gen Pc ini .
Penanda GMI_ES15_c6153_392 (Zhao et al., 2020 ) mengidentifikasi 23 galur oat yang positif untuk alel resistensi Pc39 dalam koleksi oat. Di antara galur tersebut, 16 galur memiliki hubungan silsilah dengan pembawa Pc39 yang diketahui (Gambar 2 ; Informasi Pendukung S1 ) dan 10 galur sebelumnya dianggap membawa Pc39 , seperti Dumont, Moola (AC Medallion), AC Assiniboia, Riel, AC Ronald, AC Pinnacle, Leggett, Mitika, Brusher, dan Bannister (McKenzie et al., 1983 ; Mitchell Fetch et al., 2007 ; Sowa & Paczos-Grzęda, 2020 ; Park et al., 2009, 2023 ). Percobaan fenotipe karat menggunakan 16 isolat Pca (Henningsen et al., 2024 ; Nguyen et al., 2025 ) dilakukan untuk 13 galur yang positif terhadap penanda Pc39 tetapi sebelumnya tidak dipostulatkan membawa Pc39 (Gambar S4a ; Tabel S3 ). Empat galur oat, Kowari, Forester, Drummond, dan Hokonui, menunjukkan profil resistensi yang sama terhadap diferensial Pc39 . Lima galur positif lainnya (Drover, Warrego, Culgoa, Glider, dan Possum) resisten terhadap semua isolat avirulen Pc39 ditambah satu atau lebih isolat tambahan. Dari semuanya, Drover dan Warrego keduanya memiliki hubungan silsilah dengan galur diferensial Pc39 (Gambar 2 ). Ini menunjukkan bahwa Pc39 mungkin menjadi bagian dari mekanisme resistensi pada galur-galur ini. Drover juga dipostulatkan membawa Pc91 (Nguyen et al., 2024 ). Khususnya, Glider dan Warrego sebelumnya dipostulatkan membawa Pc58 / Pc59 dan Pc61 +, masing-masing, tetapi menunjukkan profil resistensi yang sangat berbeda dari garis diferensial Pc58, Pc59, dan Pc61, yang menunjukkan bahwa Pc39 dan/atau gen lainnya, daripada gen yang dipostulatkan sebelumnya dapat berkontribusi terhadap resistensi pada garis-garis ini. Culgoa dan Possum sebelumnya dipostulatkan mengandung gen Pc yang tidak diketahui . Terakhir, empat garis oat Nugene ( PcNugene) , Genie ( Pc61+ ), Graza 80, dan Cypress menunjukkan profil resistensi yang sama terhadap Pc39 tetapi rentan terhadap setidaknya satu isolat Pca yang avirulen terhadap Pc39. Variasi ini menimbulkan ketidakpastian apakah garis-garis ini membawa gen resistensi Pc39 , meskipun semuanya positif untuk Pc39.penanda dan tiga (Nugene, Genie, dan Graza 80) berbagi koneksi silsilah dengan pembawa Pc39 yang diketahui (Gambar 2 ).
Penanda terkait Pc45 I05-0874-KOM16c1 yang diperoleh dari Gnanesh et al. ( 2015 ) dan penanda KASP_2D_181078165 yang diperoleh dari analisis asosiasi tidak positif untuk galur apa pun dalam koleksi gandum (Informasi Pendukung S1 ). Hal ini menyiratkan tidak adanya alel resistensi Pc45 dalam kultivar gandum ini.
Penanda terkait Pc54 , KASP_chr7D_46449934 , mengidentifikasi lima galur, Bambu, Diamond Black, Norway Black, Burke, dan Eagle sebagai pembawa potensial Pc54 . Akan tetapi, tidak ada hubungan silsilah yang terlihat antara galur-galur ini dan donor Pc54 (CAV1832). Selain itu, galur-galur ini juga menunjukkan profil resistensi yang berbeda terhadap galur diferensial Pc54 (Gambar S4b ), yang menunjukkan bahwa galur-galur ini tidak membawa gen resistensi ini.
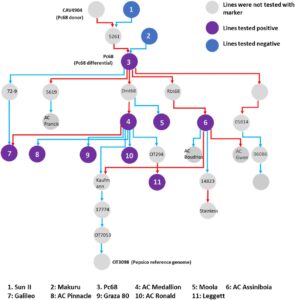
Dua penanda terkait Pc68 KASP_chr3D_466310997 dan KASP_chr3D_473653096 menunjukkan profil genotipe yang identik di seluruh rangkaian galur oat dan keduanya mengidentifikasi tujuh galur positif (Informasi Pendukung S1 ). Lima di antaranya, Leggett, AC Ronald, AC Pinnacle, Moola (AC Medalion), dan AC Assiniboia, sebelumnya dilaporkan sebagai pembawa Pc68 (Chen et al., 2006 ), sementara Galileo dan Graza 80 kemungkinan juga merupakan pembawa berdasarkan hubungan silsilahnya dengan pembawa Pc68 lainnya (Gambar 3 ) dan profil resistensi yang serupa dengan galur diferensial Pc68 (Gambar S4c ). Garis oat CDC Dancer, CDC Boyer, Starter, Makuru, Sun II, Swan, Riel, dan Ogle sebelumnya dilaporkan sebagai nonpembawa Pc68 (Chen et al., 2006 ; Kulcheski et al., 2010 ; Wong et al., 1983 ; Satheeskumar et al., 2011 ), dan mereka juga negatif untuk penanda KASP_chr3D_466310997 dan KASP_chr3D_473653096 .
4 DISKUSI
Penanda molekuler diagnostik untuk gen-gen tertentu memungkinkan pemulia untuk menggabungkan beberapa sumber resistensi yang berbeda ke dalam varietas yang sama. Hingga saat ini, hanya sejumlah kecil penanda KASP yang terkait dengan resistensi karat mahkota gandum telah divalidasi untuk digunakan dalam program pemuliaan. Studi saat ini memanfaatkan informasi pemetaan gen (Admassu-Yimer et al., 2022 ; Gnanesh et al., 2015 ; Kebede et al., 2019 ; Klos et al., 2017 ; Zhao et al., 2020 ) dan data genotipe DArTSeq (Nguyen et al., 2024 ) untuk memvalidasi penanda dan lokasi genetik untuk Pc39 , Pc45 , Pc54 , dan Pc68 dan mengembangkan penanda KASP yang lebih baik untuk penggunaan di masa mendatang dalam kegiatan pemuliaan untuk mencapai resistensi karat mahkota gandum yang tahan lama.
Penanda yang paling spesifik untuk keberadaan Pc39 , Pc45 , Pc54 , dan P68 dalam set diferensial ditemukan sebagai GMI_ES15_c6153_392 (Zhao et al., 2020 ) untuk Pc39 , I05-0874-KOM16c1 (Gnanesh et al., 2015 ) dan KASP_chr2D_181078165 (studi ini) untuk Pc45/PcKM , KASP_chr7D_464499341 (studi ini) untuk Pc54 , dan KASP_chr3D_473653096 dan KASP_chr3D_466310997 (studi ini) untuk Pc68 (Tabel 1 ). Di antara penanda yang diuji, GMI_ES01_c12570_390 ( Pc39 ) dan KASP_GMI_ES15_c15279_258 ( Pc54 ) menunjukkan heterozigositas karena sifat homoeolog dari SNP, fenomena umum pada spesies poliploid yang mengurangi efisiensi uji KASP (Mammadov et al., 2012 ). Selain itu, penanda KASP baru untuk Pc39 , yang berasal dari SNP DArTSeq kami, hanya mendeteksi genotipe SNP yang terkait dengan resistensi di semua galur yang diuji. Tantangan untuk pengembangan penanda yang sesuai untuk Pc39 kemungkinan muncul dari daerah homoeolog yang mengurangi spesifisitas primer ke kromosom tunggal atau kesalahan perakitan sekuensing dalam genom referensi OT3098 v2. Upaya masa depan untuk merancang penanda spesifik akan bermanfaat dari pemanfaatan data sekuens dari pangenom oat yang akan segera diterbitkan dan genom lain yang sudah tersedia (Avni et al., 2024 ; Kamal et al., 2022 )
Khususnya, di antara galur yang positif untuk penanda Pc39 GMI_ES15_c6153_392 , galur diferensial Pc61 (USA) dan Pc70 (USA) berkerabat dekat dengan diferensial Pc39 (Aus dan USA) pada pohon filogenetik (Nguyen et al., 2024 ). Selain itu, Pc70 (USA) diusulkan sebagai duplikat diferensial Pc39 karena mereka memiliki profil resistensi yang sama (Henningsen et al., 2024 ). Galur diferensial Pc91 dan pembawa Pc91 lainnya (HiFi dan Drover) memiliki hubungan silsilah dengan diferensial Pc39 dan semuanya diuji positif untuk penanda Pc39 . Ini selaras dengan analisis GWAS terkini dari populasi patogen karat mahkota gandum (Hewitt et al., 2023 ), yang mengidentifikasi beberapa daerah genomik di Pca yang terkait dengan virulensi pada Pc91 , salah satunya juga terkait dengan virulensi pada Pc39 . Penjelasan yang mungkin untuk ini adalah bahwa garis diferensial Pc91 dan turunannya (HiFi dan Drover) dapat membawa beberapa gen resistensi termasuk Pc39 dalam latar belakang genetiknya. Di sisi lain, Pc39 , Pc55 , dan Pc71 diusulkan menjadi alel yang berbeda dari gen yang sama berdasarkan profil resistensi yang serupa terhadap populasi Pca di Amerika Serikat dan Kanada (Chong & Seaman, 1989 ; Miller et al., 2020 ; Leonard et al., 2005 ). Selain itu, Klos et al. ( 2017 ) mengidentifikasi QTL karat mahkota yang terletak di wilayah yang sebelumnya dipetakan untuk Pc71 , selaras dengan lokasi homoeolog Pc39 (chr4C, chr5D, dan chr6A, OT3098 v2). Dalam penelitian kami, garis diferensial Pc55 dan Pc71 keduanya negatif untuk penanda Pc39 GMI_ES15_c6153_392 (Informasi Pendukung S1 ). Ini menunjukkan bahwa penanda tersebut spesifik untuk Pc39 tetapi tidak untuk Pc55 dan Pc71 . Kultivar gandum Durack sebelumnya dianggap membawa Pc39 (Park et al., 2023 ), tetapi diuji negatif untuk penanda Pc39 GMI_ES15_c6153_392 (Informasi Pendukung S1 ) dan tidak menunjukkan hubungan silsilah dengan pembawa Pc39 yang diketahui. Demikian pula, kultivar Barcoo, yang dipostulatkan membawa Pc39 , Pc61 , dan PcBett (Park, 2013 ), juga diuji negatif untuk Pc39.penanda (Informasi Pendukung S1 ) dan tidak memiliki hubungan silsilah dengan pembawa Pc39 . Lebih jauh lagi, Barcoo menunjukkan resistensi terhadap semua isolat Pca yang diuji dari Australia pada tahun 2022 dan 2023 (Henningsen et al., 2024 ; Nguyen et al., 2025 ), tidak seperti galur diferensial Pc39, Pc61, dan Bettong ( PcBett ), yang secara umum rentan terhadap isolat Pca pada tahun 2023. Hal ini menunjukkan bahwa resistensi Barcoo mungkin disebabkan oleh gen lain atau tambahan.
Sebelumnya, penanda KASP I05-0874-KOM16c1 , yang terletak pada 181.118.479 Mb pada chr2D (OT3098 v2), ditemukan terkait erat dengan Pc45 / PcKM dalam lima populasi pemetaan dan dilaporkan memiliki daya prediksi terbaik untuk ada/tidaknya Pc45 dan PcKM dalam panel 71 garis oat yang beragam (Kebede et al., 2019 ). Dalam studi ini, I05-0874-KOM16c1 dan penanda yang baru dikembangkan untuk Pc45 / PcKM , KASP_chr2D_181078165 yang berasal dari analisis asosiasi (sekitar 40Kb jauhnya dari I05-0874-KOM16c1) dalam set diferensial menunjukkan tingkat diagnostik yang tinggi secara ekuivalen, mendeteksi hanya diferensial Pc45/PcKM dalam set ini (Informasi Pendukung S1 ). Kedua penanda hanya mendeteksi alel yang terkait dengan kerentanan saat menyaring koleksi oat sebanyak 150 galur, yang menunjukkan tidak adanya Pc45 / PcKM dalam stok ini. Admassu-Yimer dkk. ( 2018 ) menunjukkan hubungan dekat antara Pc45 dan Pc53 dalam 1cM pada Mrg08 (chr2D), yang menunjukkan bahwa keduanya terkait erat atau alel. Dalam penelitian ini, garis diferensial Pc53 negatif untuk semua penanda Pc45 / PcKM (Informasi Pendukung S1 ), yang menunjukkan bahwa penanda ini membedakan antara gen-gen resistensi yang terkait ini.
Penanda KASP_chr7D_464499341 sangat diagnostik untuk Pc54 , mendeteksi hanya dua garis Pc54 yang ditunjuk dalam set diferensial (Tabel 1 ). Penanda ini dan penanda lain yang terkait dengan Pc54 (Admassu-Yimer et al., 2022 ) diidentifikasi dalam daerah duplikat tandem pada ujung distal chr7D—OT3098 v2 (Gambar S4 ). Duplikasi ini juga diamati oleh Wight et al. ( 2024 ), yang menyarankan bahwa ia mengandung gen Pc38 (dipetakan oleh Wight et al. [ 2024 ]) melalui pemetaan komparatif. Selain itu, Pc54 sebelumnya telah disarankan untuk menjadi alel atau terkait erat dengan Pc35 (Martens et al., 1980 ). Dalam penyelidikan kami saat ini, galur diferensial Pc38 dan Pc35 negatif untuk tiga dan empat penanda Pc54 , masing-masing (Informasi Pendukung S1 ). Hasil ini menunjukkan bahwa penanda tersebut secara efektif membedakan Pc54 dari dua gen Pc35 dan Pc38 . Penyaringan koleksi Oat dengan KASP_chr7D_464499341 mengungkapkan alel resistensi Pc54 menjadi langka, hanya muncul dalam lima galur oat. Namun, ini tidak menunjukkan hubungan silsilah dengan Pc54 dan profil resistensi yang sangat berbeda yang menunjukkan bahwa galur ini bukanlah pembawa Pc54 , dan mungkin telah terjadi rekombinasi antara penanda dan gen penyebab pada galur ini. Meskipun demikian, penanda ini cocok untuk seleksi Pc54 di sebagian besar latar belakang genetik yang diuji.
Penanda Pc68 KASP_avgbs_228849 , yang berasal dari GBS-SNP avgbs_228849 (Klos et al., 2017 ) , bersama dengan dua penanda yang baru dikembangkan, KASP_chr3D_466310997 dan KASP_chr3D_473653096 dari SNP DArTSeq, terletak di dalam wilayah terkait Pc68 yang sebelumnya diidentifikasi oleh Kulcheski et al. ( 2010 ), Chen et al. ( 2006 ), dan Satheeskumar et al. ( 2011 ). Wilayah ini juga merupakan bagian dari “kluster gen karat” pada chr3D yang telah dilaporkan sebelumnya, termasuk beberapa gen ketahanan karat batang (misalnya, Pc46 , Pc50 , Pc95 ) (misalnya, Fleischmann et al., 1971 ; Wong et al., 1983 ; Harder et al, 1995 ). Dalam studi ini, dua penanda KASP_chr3D_466310997 dan KASP_chr3D_473653096 spesifik untuk pembawa Pc68 dalam set diferensial dan secara akurat membedakan pembawa Pc68 yang diketahui dari yang bukan pembawa dalam koleksi oat (Informasi Pendukung S1 ). Sebaliknya, menurut Chen et al. ( 2006 ), situs bertag sekuens dan penanda SNP mereka secara tidak tepat mengidentifikasi CDC Dancer dan CDC Boyer sebagai pembawa Pc68 , meskipun garis-garis ini tidak memiliki sifat tersebut.
Khususnya, galur Graza 80 dan Galileo, yang sebelumnya tidak diduga memiliki Pc68, juga positif terhadap dua penanda Pc68 baru dan menunjukkan profil resistensi yang serupa terhadap pembawa Pc68. Kedua galur tersebut merupakan keturunan AC Medallion ( pembawa Pc68 yang diketahui ), yang menunjukkan bahwa keduanya kemungkinan juga membawa Pc68 . Dua penanda KASP yang baru dikembangkan untuk Pc68 dianggap sebagai diagnostik untuk seleksi Pc68 .
Sebagai kesimpulan, analisis asosiasi kami dalam set diferensial karat mahkota gandum mengonfirmasi lokasi fisik Pc39 , Pc45 / PcKM , Pc54 , dan Pc68 . Penanda KASP yang dikaitkan dengan gen resistensi, yang dikembangkan dari penelitian ini dan penelitian sebelumnya, divalidasi sebagai diagnostik yang tinggi untuk gen Pc yang ditentukan dan mengidentifikasi beberapa pembawa potensial Pc39 dan Pc68 dalam koleksi gandum. Hasil penelitian ini menawarkan alat yang berharga bagi MAS dalam program pemuliaan untuk memfasilitasi integrasi lokus resistensi ini ke dalam varietas gandum dan pembuatan galur dengan kombinasi gen resistensi karat mahkota.




Tinggalkan Balasan