ABSTRAK
Sel ovarium hamster Cina (CHO) merupakan inang utama untuk produksi biofarmasi. Untuk memenuhi permintaan yang semakin meningkat akan produktivitas, kualitas, dan ekspresi molekul yang kompleks, rekayasa genetika, khususnya clustered regular interspaced short palindromic repeats (CRISPR)-mediated gene knockout (KO), digunakan secara luas untuk mengoptimalkan kinerja sel inang. Namun, penyaringan sistematis target KO tetap menjadi tantangan karena proses pembuatan dan evaluasi klon individual yang padat karya. Dalam studi ini, kami menyajikan alur kerja CRISPR yang kuat dan berthroughput tinggi menggunakan kumpulan KO yang stabil dalam sel CHO. Kumpulan ini mempertahankan stabilitas genetik selama lebih dari 6 minggu, termasuk dalam konfigurasi multipleks yang menargetkan hingga tujuh gen secara bersamaan. Dibandingkan dengan pendekatan klonal, kumpulan KO mengurangi variabilitas yang disebabkan oleh heterogenitas klonal dan lebih mencerminkan fenotipe populasi sel inang. Kami menunjukkan kegunaan pendekatan ini dengan mereproduksi efek fenotipe yang menguntungkan dari fibronectin 1 (FN1) KO, khususnya durasi kultur yang diperpanjang dan viabilitas tahap akhir yang ditingkatkan dalam proses fed-batch. Alur kerja ini memungkinkan identifikasi dan evaluasi target KO yang menjanjikan secara efisien tanpa perlu membuat dan menguji sejumlah besar klon. Secara keseluruhan, proses penyaringan meningkat 2,5 kali lipat dan jangka waktu dipadatkan dari 9 menjadi 5 minggu. Hal ini memberikan alternatif yang dapat diskalakan dan efisien untuk penyaringan klon tradisional, yang mempercepat penemuan untuk rekayasa lini sel CHO untuk pengembangan biofarmasi.
Singkatan
Kas
Protein terkait CRISPR
MEMILIH
ovarium hamster cina
Bahasa Indonesia: CRISPR
pengulangan palindromik pendek yang berkelompok dan berjarak teratur
DNA
asam desoksiribonukleat
FITC-LCA
fluorescein isothiocyanate lensa berlabel agglutinin culinaris
FN1
fibronektin 1
FUT8
α1,6-fukosiltransferase 8
DIA
efisiensi tinggi
Dalam Bahasa Del
penyisipan/penghapusan
KO
pukulan knockout
AKU
efisiensi rendah
LPL
lipoprotein lipase
PCOLCE
prokolagen C-endopeptidase penambah
PXDN1
peroksidase 1
RNA
asam ribonukleat
RNP
ribonukleoprotein
RNA-sg
panduan tunggal RNA
THBS1
trombospondin 1
VCC
konsentrasi sel yang layak
1 Pendahuluan
Meskipun ada kemajuan signifikan dalam pembuatan biofarmasi menggunakan sel ovarium hamster Cina (CHO) selama beberapa dekade terakhir [ 1 ], ada permintaan ekonomi yang kuat untuk strategi yang meningkatkan produktivitas bioproses, meningkatkan stabilitas garis sel, dan memungkinkan rekayasa sifat-sifat yang diinginkan tertentu dalam sel CHO [ 2 – 6 ]. Penemuan sistem clustered regular interspaced short palindromic repeats (CRISPR)/CRISPR-associated protein (Cas)9 sebagai alat penyuntingan gen selain metode analitis dalam transkriptomik, metabolomik, dan proteomik telah memungkinkan wawasan yang belum pernah terjadi sebelumnya ke dalam cara kerja internal garis sel produksi, membuat rekayasa garis sel inang superior menjadi layak. Manipulasi genetik telah terbukti secara efektif mengurangi apoptosis [ 2 , 7 ], meningkatkan stabilitas produk [ 8 , 9 ], dan menghilangkan fucosilasi antibodi [ 10 ]. Studi lebih lanjut telah menunjukkan peningkatan produktivitas dan kapasitas sekresi sel CHO [ 11 – 13 ] dan mengurangi kadar protein sel inang [ 14 , 15 ] setelah perubahan genetik. Meskipun generasi garis sel inang yang direkayasa telah menjadi lebih umum, penyaringan padat karya yang diperlukan untuk mengidentifikasi klon sel tunggal yang diedit dengan benar secara signifikan membatasi jumlah klon yang disaring dalam studi yang menyelidiki efek modifikasi genetik. Keterbatasan ini terutama terlihat dalam studi multipleks di mana beberapa suntingan gen diperlukan. Misalnya, mengisolasi klon knockout dekaplex (KO) sel tunggal memerlukan penyaringan sekitar 704 klon untuk akhirnya mengidentifikasi hanya enam klon KO yang berhasil diedit [ 16 ]. Untuk menghasilkan sel KO multigen, seseorang dapat mengurangi jumlah gen yang ditangani dalam transfeksi tunggal dan menambahkan beberapa langkah subkloning. Untuk tujuan ini, beberapa penelitian telah menunjukkan pendekatan multipleks yang efektif untuk KO beberapa gen dalam satu sel untuk mengurangi profil protein sel inang [ 15 ] dengan mengurangi jumlah gen yang dihilangkan per putaran dan melakukan putaran pengeditan dan kloning berurutan.
Selain itu, menyelidiki sejumlah kecil klon sel tunggal sebagai proksi untuk mengidentifikasi efek rekayasa genetika memiliki keterbatasan yang penting. Kloning sel tunggal memperkenalkan heterogenitas yang signifikan dalam populasi yang diturunkan [ 17 – 21 ]. Sel CHO menunjukkan genom aneuploid tidak stabil yang melekat dengan penataan ulang genom yang sering, perubahan dalam polimorfisme nukleotida tunggal, dan variasi jumlah salinan yang signifikan [ 22 ]. Selain itu, selama kloning sel tunggal, sel-sel individual dapat mengalami penataan ulang genom yang substansial [ 20 ] dan perubahan dalam metilasi asam desoksiribonukleat (DNA) [ 17 , 23 ], yang pada gilirannya juga memengaruhi karakteristik klon. Banyak penelitian bergantung pada sejumlah kecil klon KO, seringkali kurang dari 10–15, terkadang bahkan hanya klon KO tunggal untuk menyaring efek KO. Klon-klon yang secara inheren sangat bervariasi ini digunakan untuk perbandingan dengan klon tipe liar (WT) yang juga sangat bervariasi. Ukuran sampel yang terbatas ini meningkatkan risiko hasil yang bias dan menantang reproduktifitas temuan, terutama karena variabilitas klonal yang tinggi yang tampak pada sel CHO. Tingkat heterogenitas yang tinggi pada klon sel tunggal ini mempersulit penilaian yang andal terhadap perubahan fenotip yang disebabkan KO atau ketiadaan perubahan tersebut. Selain itu, melakukan putaran penyuntingan dan kloning secara berurutan menimbulkan bias seleksi yang inheren, di mana pilihan klon awal memberikan efek pendiri yang menyebar melalui putaran rekayasa dan penyaringan berikutnya.
Meskipun demikian, sebagian besar penelitian menyelidiki jumlah klon yang tidak mencukupi untuk menilai secara akurat efek KO pada perubahan perilaku bioproses sel. Di sini, kami menyajikan sistem penyaringan baru untuk mengevaluasi efek KO secara langsung pada tahap pool, mencapai efisiensi KO yang tinggi dalam pool KO heterogen melalui beberapa transfeksi dan menunjukkan stabilitas genotipe dan fenotipik dari pool KO heterogen. Dengan menggunakan sistem ini, kami mengonfirmasi efek menguntungkan yang sebelumnya diidentifikasi (tidak dipublikasikan) dari fibronectin 1 (FN1) KO sebagai modifikasi genetik yang meningkatkan durasi bioproses, dengan titer akhir meningkat hingga dua kali lipat dan perilaku pertumbuhan bioproses yang ditingkatkan dalam bioproses fed-batch shake flask.
2 Metode
2.1 Kultur Sel
Sel CHO DG44 diperoleh dari Lawrence Chasin (Columbia University, NY) dan diadaptasi secara in-house untuk pertumbuhan suspensi bebas serum dalam medium yang ditentukan secara kimia. Sel-sel tersebut dikultur dalam medium CD DG44 bebas serum dan antibiotik (Gibco, Carlsbad, CA, AS) yang dilengkapi dengan glutamin 6 mM (Sartorius Stedim Cellca GmbH, Ulm, Jerman) dan 0,001% Pluronic F-68 (Gibco, Carlsbad, CA, AS). Sel CHO dikultur dalam 50 mL dalam labu kocok 250 mL (Corning, NY, AS) yang dilewatkan setiap 2 hari hingga kepadatan inokulasi 2E5 sel/mL. Setelah transfeksi, sel dikulturkan dalam 1 mL dalam pelat 24-sumur (Nunc, Roskilde, Denmark) dan setelah pertumbuhan sel mencukupi, sel diekspansi hingga 4 mL dalam 6-sumur (Nunc, Roskilde, Denmark) dan kemudian hingga 25 mL dalam labu kocok 125 mL (Corning, NY, AS) untuk penyimpanan sel dan eksperimen lebih lanjut. Semua kultur, kecuali pelat 24-, 96-, dan 384-sumur, dikocok pada kecepatan linier 110 rpm dan dikulturkan dalam kelembapan 80% pada suhu 36,8°C dan pCO 2 7,5% kecuali dinyatakan lain (kondisi standar). Penghitungan sel untuk kultur dan ekspansi sel rutin dilakukan menggunakan CASY Cell Counter & Analyzer (OMNI Life Science, Bremen, Jerman).
2.2 Desain dan Penyaringan gRNA Sintetis
Target gen untuk penelitian ini ditunjukkan pada Tabel 1. Urutan RNA pemandu dirancang menggunakan perangkat lunak Geneious Prime, versi 11.0.11 (Biomatters, Auckland, Selandia Baru), atau perangkat lunak CRISPR Guide RNA Design ( https://www.benchling.com ) yang menargetkan ekson awal yang ada di semua varian transkrip untuk menginduksi mutasi frameshift awal, yang membuat protein yang diterjemahkan tidak berfungsi. gRNA dipilih berdasarkan penilaian efisiensi pada target [ 24 ] dan spesifisitas di luar target [ 25 ]. Dipastikan bahwa kemungkinan di luar target berada dalam urutan non-pengkodean protein dari genom inang. Penilaian di luar target dilakukan terhadap rakitan genom milik sendiri, yang diurutkan sepenuhnya, dan diberi anotasi dari garis sel inang (Sartorius Stedim Cellca GmbH). Tiga RNA pemandu tunggal (sgRNA) disaring untuk setiap gen target untuk menggunakan sgRNA efisiensi tertinggi dalam eksperimen hilir. sgRNA dipesan sebagai TrueGuide Snythetic gRNA di ThermoFisher Scientific (Thermo Fisher Scientific, Waltham, MA, AS) dan ditransfeksi dengan TrueCut Cas9 Protein v2 (Thermo Fisher Scientific) dalam rasio 1:1 sebagai 7,5 pmol ribonucleoprotein (RNP) yang telah dirakit sebelumnya ke dalam sel 2E5 menggunakan Sistem Transfeksi NEON dan Kit Sistem Transfeksi NEON 10 µL (Thermo Fisher Scientific). Parameter transfeksi ditetapkan ke 1700 V, lebar pulsa 20 ms, dan 1 pulsa. Sel inang yang ditransfeksi RNP dikenakan genotyping dengan ekstraksi DNA genomik, PCR, dan analisis sekuensing Sanger diikuti oleh analisis Inference of CRISPR Edits (ICE) 48 jam setelah transfeksi seperti yang dijelaskan di bagian 2.3 .
| Gen | Nama lengkap | Fungsi/peran | ID UniProt |
|---|---|---|---|
| FUT8 | Fukosiltransferase 8 | Terlibat dalam fucosylation, proses glikosilasi utama | G3HCE4 |
| FN1 | Fibronektin 1 | Protein ECM terlibat dalam adhesi dan migrasi sel | A0A3L7IDB0 |
| LPL | Lipoprotein lipase | Mengkatalisis hidrolisis trigliserida | A0A3L7IKX6 |
| NID1 | Nidogen 1 | Menghubungkan laminin dan kolagen di ECM | A0A3L7HVW3 |
| PCOLCE | Penguat Prokolagen C-endopeptidase | Mengatur pemrosesan kolagen dan remodeling ECM | G3I664 |
| Bahasa Indonesia: PXDN | Peroksidase | Terlibat dalam pembentukan ECM dan pensinyalan H 2 O 2 | A0A3L7HCC3 |
| THBS1 | Trombospondin 1 | Menengahi interaksi sel ke sel dan sel ke matriks | G3HHV4 |
| BGN | Biglycan | Proteoglikan kecil yang terlibat dalam struktur ECM | A0A061HUR7 |
Catatan : Yang ditampilkan adalah simbol gen, nama lengkap, fungsi, dan ID UniProt.
2.3 Genotipe Kumpulan dan Klon
DNA genomik diekstraksi dari kumpulan transfeksi atau klon sel tunggal menggunakan QuickExtract DNA Extraction Solution (Lucigen), diikuti oleh PCR untuk mengamplifikasi daerah genomik yang mengapit situs pemotongan sgRNA yang bertujuan untuk 200–300 pasangan basa ke hulu dan hilir. Amplikon dikirim untuk pemurnian PCR dan pengurutan Sanger di Microsynth Seqlab GmbH (Göttingen, Lower Saxony, Jerman). Jejak pengurutan Sanger untuk setiap sampel uji (yang diedit) dan sampel kontrol yang sesuai (yang tidak diedit) diunggah ke perangkat lunak ICE, versi 3.0 (ice.synthego.com) (Synthego, Menlo Park, AS) dan dianalisis sesuai dengan petunjuk pengembang. Analisis ICE memberikan persentase penyisipan/penghapusan (InDel) dan skor KO. Persentase InDel mencerminkan efisiensi pengeditan jejak yang diedit dibandingkan dengan jejak kontrol, terlepas dari apakah InDel menghasilkan frameshift. Skor KO menunjukkan efisiensi KO, yang mewakili proporsi sel dengan InDel frameshift atau penghapusan fragmen, yang kemungkinan menghasilkan KO fungsional. gRNA dengan skor KO tertinggi untuk target gen tertentu dipilih untuk efisiensi penyuntingan tinggi dan profil InDel yang baik, yang dicirikan oleh InDel yang sebagian besar berada di luar bingkai dan skor KO tinggi, dengan preferensi untuk sgRNA yang menghasilkan berbagai InDel terbatas, idealnya satu jenis dominan.
2.4 Pembuatan Klon KO dan Kumpulan Proporsi KO Tinggi
Untuk menghasilkan populasi KO dari sel inang CHO DG44 untuk semua gen target, transfeksi dilakukan menggunakan sgRNA terbaik yang dipilih sebelumnya dari tiga sgRNA yang disaring. Kumpulan RNP-transfeksi tunggal ini kemudian digunakan untuk kloning sel tunggal untuk mendapatkan klon KO untuk eksperimen lebih lanjut. Klon sel tunggal dihasilkan menggunakan kloning berbasis nanowell berthroughput tinggi yang diverifikasi gambar dengan CellCelector (Sartorius ALS GmbH, Jena, Jerman). Setiap sumur dari pelat 24 nanowell diisi dengan media kloning 0,5 mL milik sendiri. Pelat disentrifugasi (800 x g , 5 menit) untuk sepenuhnya mengendapkan media ke dalam kisi bawah nanowell untuk memastikan kontak cairan penuh. Sebanyak 2000 sel disemai ke dalam sumur ini dengan mengencerkan sel target menjadi 4000 sel/mL dalam media kloning dan menaburkan 0,5 mL pengenceran ini ke dalam satu nanowell. Sebelum penyemaian, sampel dilewatkan melalui saringan sel (Corning, NY, AS). Masing-masing dari 24 sumur berisi sekitar 4400 nanowell, sehingga menghasilkan <0,5 sel yang disemai per sumur. Pada akhir penyemaian, pelat disentrifugasi lagi (300 x g , 3 menit) untuk mengendapkan semua sel. Sel dipindai menggunakan CellCelector (Sartorius ALS GmbH) pada hari penyemaian untuk memastikan monoklonalitas dan kemudian lagi setelah 4 hari untuk menilai pertumbuhan dan memilih klon yang tumbuh lebih besar (15–20 sel) ke dalam pelat 384 sumur yang berisi media kultivasi standar. Konfluensi klon diukur menggunakan CellMetric (Solentim, Bournemouth, Inggris) secara berkala dari Hari ke-7 hingga ke-14. Setelah sebagian besar klon mencapai konfluensi 70%–100%, semua klon dipindahkan ke pelat 96 sumur dan dilewatkan setiap 3–4 hari saat genotipe berlangsung. Klon KO lengkap diperluas ke pelat 12-sumur dalam media kultur standar 1,5 mL sebelum diperluas ke tabung 6-sumur dan tabung goyang dalam kondisi standar. Untuk menghasilkan kumpulan KO dengan skor KO tinggi, transfeksi RNP yang sama dilakukan tiga kali berurutan dengan jarak 48 jam, dengan sel yang sama. Kumpulan ini kemudian diperluas untuk eksperimen lebih lanjut.
2.5 Uji FITC-LCA untuk Analisis Fungsional Kumpulan dan Klon KO FUT8
Untuk menilai hasil fenotipik transfeksi RNP yang menargetkan gen α1,6-fucosyltransferase 8 (FUT8), pool diwarnai dengan fluorescein isothiocyanate berlabel lens culinaris agglutinin (FITC-LCA), yang mengikat residu fucose pada protein membran. LCA sebelumnya juga telah digunakan untuk memperkaya subpopulasi KO FUT8 secara selektif [ 26 ]. Campuran sel FITC-positif dan FITC-negatif diharapkan dalam pool yang diedit CRISPR, dengan sel FUT8 KO menunjukkan peralihan lengkap ke populasi FITC-negatif. Pengujian ini digunakan untuk memvalidasi hasil dari analisis ICE sebagai metode ortogonal, yang memungkinkan pengukuran hasil fenotipik selain hasil genetik. Pengujian ini digunakan untuk menilai efisiensi KO dari sgRNA penargetan FUT8 yang berbeda pada tingkat pool. Sel host CHO DG44 yang tidak ditransfeksi digunakan sebagai kontrol negatif. Sel disiapkan dengan sentrifugasi (390 x g , 3 menit) dan pencucian dua kali dengan buffer CASYton dingin (OMNI Life Science, Bremen, Jerman). Pewarnaan dilakukan dengan menginkubasi sel dengan 5 µg/µL FITC-LCA (Thermo Fisher Scientific) selama 30 menit pada suhu 4°C, diikuti oleh dua siklus pencucian tambahan. Fluoresensi diukur dengan eksitasi pada 488 nm menggunakan IntelliCyte iQue Screener (Sartorius, Göttingen, Jerman).
2.6 Karakterisasi Fed-Batch dari KO Pools dan Clone
Untuk mengevaluasi perubahan kinerja bioproses dari kumpulan dan klon KO, percobaan fed-batch dilakukan dengan menginokulasi 25 mL 4Cell SmartCHO PM (Sartorius, Ulm, Jerman) yang disuplemen dengan 6 mM glutamin (Sartorius, Ulm, Jerman) dalam labu kocok 125 mL (Corning, NY, AS) hingga 3E5 sel hidup/mL. Kultur diberi makan dengan 4Cell SmartCHO FMA (Sartorius, Ulm, Jerman) dan 4Cell SmartCHO FMB (Sartorius, Ulm, Jerman). Konsentrasi glukosa disesuaikan menjadi 5 g/L sebagai tambahan pemberian makan. Setelah 14 hari atau setelah viabilitas mencapai di bawah 70%, kultur dipanen untuk mengumpulkan supernatan.
Titer antibodi diukur menggunakan sistem Octet HTX (Sartorius, Göttingen, Jerman). Konsentrasi antibodi dalam sampel retensi dihitung menggunakan kurva standar antibodi yang dimurnikan.
2.7 Perhitungan Daya Statistik
Untuk menentukan ukuran sampel yang diperlukan ( n ) untuk variasi tertentu dalam parameter proses, kami menghitung ukuran sampel berdasarkan daya 90% dengan tingkat signifikansi α = 0,05 untuk perbedaan rata-rata tertentu, disebut ukuran efek (ES), menggunakan Persamaan ( 1 ). Skor Z dihitung di Excel menggunakan fungsi invers normal standar (NORM.S.INV) [ 27 , 28 ].
Persamaan ( 1 ) Perhitungan ukuran sampel.

3 Hasil dan Pembahasan
3.1 Melewati Kloning Sel Tunggal untuk Penyaringan Efek KO dalam Kumpulan Menghemat Waktu 5–6 Minggu
Skrining KO tradisional dalam sel CHO biasanya memerlukan kloning sel tunggal, sebuah proses yang melelahkan yang memerlukan beberapa minggu kultivasi dan skrining [ 16 , 15 ]. Dengan kemajuan alat CRISPR, pendekatan tergabung untuk skrining telah menjadi lebih umum. Di sini, pustaka dari sekuens sgRNA yang berbeda dikirimkan ke dalam sel, menghasilkan kumpulan heterogen yang kemudian dikenakan proses seleksi fenotipik atau fungsional. Sebagai pembacaan, distribusi jumlah sgRNA ditangkap oleh NGS [ 29 ]. Pendekatan tergabung ini juga telah ditunjukkan dalam skrining interferensi RNA [ 30 ]. Di sini, kami bertujuan untuk mentransfer prinsip penggunaan kumpulan untuk tujuan skrining ke proses platform KO tradisional kami. Seperti yang ditunjukkan pada Gambar 1 , proses ini mencakup beberapa langkah dari penyemaian sel hingga perluasan klon, yang secara signifikan menunda penilaian fenotipik. Dengan menyaring efek KO secara langsung dalam kumpulan KO heterogen, kebutuhan untuk kloning sel tunggal dihilangkan, mengurangi jangka waktu untuk menghasilkan populasi KO yang dapat digunakan untuk studi fungsional hilir dari 4 hingga 5 minggu menjadi hanya 1 minggu.
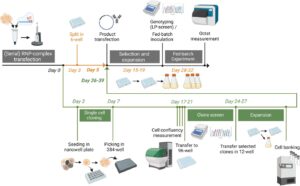
Untuk memungkinkan hal ini, efisiensi KO yang tinggi harus dicapai dalam populasi gabungan, dan hubungan linear antara genotipe dan fenotipe serta stabilitas genotipe di seluruh proses yang dilakukan harus ditunjukkan. Pendekatan semacam itu tidak hanya mempercepat identifikasi fenotipe KO tetapi juga mengurangi tuntutan tenaga kerja dan sumber daya. Pendekatan ini sangat berguna dalam studi penyuntingan gen multipleks, yang memungkinkan identifikasi awal yang efisien terhadap kombinasi gen KO yang berdampak.
3.2 Korelasi Genotipe dan Fenotipe dalam Kumpulan KO Fucosyltransferase 8 yang Heterogen
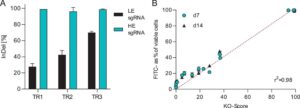
Mencapai efisiensi KO yang tinggi sangat penting untuk menyaring kandidat KO secara efektif untuk perubahan perilaku bioproses dalam kumpulan KO heterogen. Ini diperlukan untuk membatasi efek masking dari subpopulasi WT yang ada dalam kumpulan KO. Transfeksi berurutan digunakan untuk meningkatkan efisiensi sgRNA efisiensi rendah dan tinggi yang menargetkan gen FUT8, yang pada akhirnya mencapai peningkatan efisiensi KO yang signifikan setelah tiga transfeksi berurutan. Metode ini sebelumnya telah dijelaskan dalam konteks KO dekaplex gen sel CHO [ 16 ]. Gen FUT8 memungkinkan identifikasi cepat subpopulasi yang tidak mengekspresikan FUT8 fungsional dengan menganalisis proporsi sel yang menampilkan protein membran nonfucosilasi [ 10 ]. Gen model yang umum dipelajari ini mengkatalisis penambahan fucose ke antibodi IgG1 dalam sel CHO. Menghilangkan FUT8 mengurangi sitotoksisitas yang dimediasi sel yang bergantung pada antibodi [ 31 – 33 ]. Gambar 2A menunjukkan peningkatan persentase InDel untuk sgRNA efisiensi rendah (LE) dan efisiensi tinggi (HE) setelah satu, dua, dan tiga transfeksi berurutan. Transfeksi penyuntingan gen dilakukan menggunakan kompleks RNP yang sama pada interval 48 jam. SgRNA HE mencapai frekuensi InDel 99% ± 0% setelah transfeksi tunggal, sedangkan sgRNA LE menunjukkan peningkatan dari 28,0% ± 3,8% setelah transfeksi pertama menjadi frekuensi InDel 70,0% ± 1,6% setelah tiga transfeksi. Kumpulan yang dihasilkan menunjukkan stabilitas genotipe dari waktu ke waktu, tanpa perubahan yang terdeteksi dalam profil InDel selama 2 minggu kultivasi (Gambar 2B ). Skor KO dan data fenotipik yang ditunjukkan pada Gambar 2B masing-masing sesuai dengan kumpulan sel yang dipanen pada Hari ke-7 dan ke-14 setelah transfeksi akhir dari setiap seri transfeksi berurutan.
3.3 Kolam KO Heterogen Stabil dalam Pendekatan Tunggal dan Multipleks
Untuk menyelidiki lebih lanjut stabilitas kumpulan KO heterogen, empat gen tambahan dievaluasi dalam pendekatan singleplex. Sel CHO menjalani tiga transfeksi berurutan, kumpulan tersebut dikultur selama 4 minggu dan digenotipe 1 dan 4 minggu pascatransfeksi. Transfeksi dengan kompleks RNP yang menargetkan gen lipoprotein lipase (LPL), PRDX1, dan trombospondin 1 (THBS1) menunjukkan skor KO > 80% setelah transfeksi tunggal dengan transfeksi tambahan yang tidak meningkatkan skor lebih lanjut. Kumpulan transfeksi yang menangani gen FN1 menunjukkan peningkatan yang nyata dari 36,5% ± 14,9% menjadi 80,5% ± 3,5% setelah tiga transfeksi. Skor KO untuk LPL, PRDX1, dan THBS1 tidak meningkat dengan transfeksi berikutnya, kemungkinan karena pengeditan yang hampir lengkap dari semua sekuens WT setelah transfeksi awal. Viabilitas sel tetap di atas 90% selama proses transfeksi berurutan, tanpa penurunan yang terdeteksi setelah putaran transfeksi apa pun (data tidak ditampilkan), konsisten dengan laporan sebelumnya [ 16 ]. Semua kelompok menunjukkan stabilitas dalam skor KO, mempertahankan skor KO awal selama jangka waktu minimal 4 minggu (Gambar 3A ).
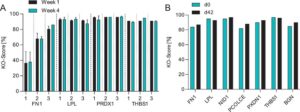
Kumpulan CHO KO yang dihasilkan menunjukkan stabilitas baik dalam pendekatan tunggal maupun multipleks. Stabilitas dan linearitas korelasi genotipe-fenotipe dapat bervariasi tergantung pada gen target.
3.4 Kumpulan CHO KO Lebih Cocok Dibandingkan Klon untuk Menghubungkan Genotipe dengan Fenotipe
Gen FN1, yang sebelumnya kami identifikasi memiliki efek peningkatan bioproses pada fenotipe sel (tidak dipublikasikan), dipilih untuk membandingkan pendekatan berbasis klon sel tunggal tradisional dengan alur kerja berbasis kumpulan (Gambar 1 ). FN1 adalah bagian dari matriks ekstraseluler dan memainkan peran kunci dalam adhesi sel melalui interaksi dengan integrin dan pensinyalan hilir [ 35 ]. Dengan tidak adanya FN1, pensinyalan adhesi yang dimediasi integrin dapat berkurang dan mengurangi pembentukan agregat sel melalui interaksi sel-ke-sel [ 36 , 37 ]. Selain adhesi sel, FN1 juga mengatur migrasi sel, proliferasi, dan kelangsungan hidup dengan pensinyalan melalui jalur pensinyalan PI3K/Akt, MAPK/ERK [ 38 ]. Tidak adanya FN1 melalui KO dapat berkontribusi pada peningkatan kinerja kultur dengan menunda respons stres yang mengarah pada peningkatan agregasi sel dan apoptosis. Bahasa Indonesia: Untuk memvalidasi pool-platform kami dan mereproduksi temuan ini, kami membuat pool KO dan klon KO sel tunggal dari lini sel WT CHO DG44 internal menggunakan kompleks RNP yang menargetkan ekson awal gen FN1. Secara total, 96 klon sel tunggal dibuat untuk skrining genotipe yang 12 klonnya dipilih berdasarkan genotipe KO dan pertumbuhan klonalnya. Klon dipilih berdasarkan keberadaan InDel yang menginduksi frameshift biallelic (yaitu, insersi atau delesi yang tidak habis dibagi tiga) di wilayah target, sebagaimana ditentukan oleh sekuensing Sanger dan analisis ICE, di samping pertumbuhan klonal yang kuat. Pool KO FN1 dibuat dengan kompleks RNP yang sama yang digunakan untuk pembuatan klon KO dengan tiga transfeksi berurutan untuk mencapai skor KO yang tinggi. Untuk mengonfirmasi bahwa KO FN1 mengurangi ekspresi protein, Western blot dilakukan pada pool KO yang dibuat menggunakan alur kerja KO yang sama. Tingkat FN1 relatif, dinilai dengan intensitas pita yang dinormalkan ke ACTB, menunjukkan pengurangan rata-rata 52% dalam kumpulan KO, yang mendukung gangguan tingkat protein yang efektif. Ekspresi residual konsisten dengan skor KO di bawah 100%, karena beberapa alel mungkin tetap tidak diedit atau heterozigot (Gambar S1A, B ). Kumpulan/klon KO dan kumpulan WT digunakan untuk menghasilkan kumpulan produksi stabil yang mengekspresikan tiga model antibodi IgG1 (NISTmAb, IgG1 A, dan IgG1 B) melalui seleksi dihidrofolat reduktase (DHFR) (Institut Nasional Standar dan Teknologi [NIST] [ 39]). Kumpulan produksi ini kemudian diperluas untuk evaluasi fed-batch shake-flask. Eksperimen fed-batch shake-flask dilakukan secara simultan dengan klon KO, kumpulan KO, dan kumpulan WT untuk masing-masing dari tiga molekul IgG1. Selama bioproses fed-batch, baik klon KO maupun kumpulan KO menunjukkan penurunan viabilitas yang tertunda, yang mengakibatkan durasi proses yang lebih lama dan viabilitas yang lebih tinggi, yang berhubungan dengan peningkatan konsentrasi sel viabel integral (IVCC), yang pada akhirnya juga mengarah ke titer yang lebih tinggi (Gambar 4A, C, D ). Kumpulan KO menunjukkan dinamika pertumbuhan yang serupa dengan WT hingga puncak konsentrasi sel viabel (VCC) pada Hari ke-7, diikuti oleh fase stasioner yang diperpanjang dengan viabilitas sel yang tinggi. Efek ini juga diamati pada klon KO. Namun, puncak VCC sangat bervariasi. Puncak VCC yang lebih rendah kemungkinan dapat dikaitkan dengan variasi klonal yang diperkenalkan selama isolasi sel tunggal, serta perbedaan viabilitas awal. Misalnya, dalam kasus IgG1 A, beberapa klon KO menunjukkan viabilitas awal serendah ∼80%, dibandingkan dengan kumpulan KO FN1 dan kontrol WT. Meskipun terdapat variabilitas ini, klon KO mempertahankan viabilitas yang diperpanjang hingga akhir proses, dengan banyak replikasi menunjukkan viabilitas lebih tinggi dari 70% bahkan pada Hari ke-14 dari proses fed-batch. Dengan demikian, pergeseran karakteristik proses dipertahankan secara konsisten di kedua klon KO dan kumpulan KO, yang menunjukkan reproduktifitas yang kuat di berbagai molekul dan eksperimen IgG1. Efek peningkatan produktivitas dari KO FN1 hanya direproduksi sebagian dalam klon KO tetapi lebih nyata dalam kumpulan KO, yang mempertahankan heterogenitas penuh populasi inang dan kurang rentan terhadap bias dari variasi genetik atau fenotipik acak yang dapat muncul selama kloning sel tunggal. Kumpulan KO heterogen menunjukkan peningkatan titer akhir 1,4 hingga 2,1 kali lipat untuk molekul IgG1 yang diuji. Khususnya, peningkatan ini sebagian tidak ada pada klon KO, kemungkinan karena variasi latar belakang sel yang melekat, yang mempersulit reproduksibilitas efek pada klon individual. Sebaliknya, kumpulan KO menangkap heterogenitas penuh populasi sel inang, yang berfungsi sebagai proksi yang lebih akurat untuk menilai perubahan di seluruh kumpulan sel inang. Dengan melewati kloning sel tunggal, yang dapat menimbulkan variabilitas dan gangguan, pendekatan ini menawarkan keuntungan substansial untuk menyaring gen KO kandidat secara efisien. Selain itu, stabilitas genetik kumpulan KO dipantau. Skor KO tetap tinggi secara konsisten (>80%) untuk klon KO dan kumpulan KO selama transfeksi, seleksi, dan bioproses fed-batch (Gambar 4B)). Stabilitas genetik melalui proses penyaringan KO dan evaluasi fed-batch ini sangat penting untuk mempertahankan fenotipe yang diinginkan untuk penemuan target putatif. Temuan ini menggarisbawahi keuntungan menggunakan kumpulan KO untuk tujuan penyaringan. Heterogenitas kumpulan tampaknya menangkap rentang fenotipe yang lebih luas, yang berpotensi mengungkapkan peningkatan yang mungkin terlewatkan saat menggunakan klon individual. Untuk memanfaatkan klon secara efisien guna membuat estimasi akurat tentang perubahan signifikan dalam titer akan memerlukan penggunaan klon dalam jumlah yang sangat tinggi. Lebih jauh, penggunaan kumpulan dapat mengurangi gangguan dalam data dan meningkatkan hasil dalam proses penyaringan, seperti yang ditunjukkan dalam tantangan dan peningkatan yang disebutkan sebelumnya. Peningkatan produktivitas yang konsisten yang diamati di seluruh protein bioterapi yang berbeda menunjukkan bahwa KO FN1 mungkin memiliki penerapan yang luas dalam meningkatkan kinerja sel CHO untuk berbagai produk.
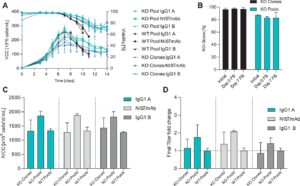
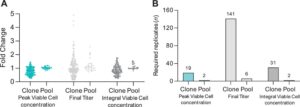
Penggunaan pool KO dalam studi penyaringan genetik menawarkan keuntungan signifikan dibandingkan pendekatan klonal tradisional. Ketika membandingkan variabilitas empiris klon dan pool dalam eksperimen fed-batch kami di seluruh parameter bioproses utama: VCC puncak, titer akhir, dan IVCC, klon dan pool berperilaku berbeda (Gambar 5A ). Populasi klonal menunjukkan heterogenitas yang cukup besar di seluruh metrik kinerja utama ini, sebagaimana dibuktikan oleh berbagai titik data untuk klon di ketiga parameter. VCC puncak dalam populasi klonal bervariasi dari perubahan 0,26 hingga 1,45 kali lipat dibandingkan dengan WT masing-masing, sementara populasi pool menunjukkan kisaran yang lebih sempit yaitu 0,83–1,22. Demikian pula, titer akhir berkisar antara perubahan 0,12 hingga 4,91 kali lipat pada klon, sementara pool menunjukkan kisaran yang lebih konsisten yaitu 0,74–2,57. Bahasa Indonesia: Untuk IVCC, klon menunjukkan rentang perubahan lipatan 0,24–1,77, sedangkan kumpulan menunjukkan rentang yang lebih terbatas dari 0,81 hingga 1,53. Variabilitas substansial yang diamati dalam populasi klon mempersulit penilaian modifikasi genetik yang akurat, karena memilih sejumlah klon yang terbatas dapat menghasilkan data yang tidak mewakili keseluruhan populasi. Sebaliknya, kumpulan KO menunjukkan variabilitas yang sangat berkurang, dengan pengelompokan titik data yang lebih ketat—terutama dalam titer akhir—yang memungkinkan pandangan yang lebih andal tentang dampak modifikasi genetik. Pendekatan kumpulan ini menangkap representasi yang lebih luas dari populasi sel yang diedit, menghilangkan bias dan gangguan yang diperkenalkan oleh pemilihan klon tunggal. Variabilitas tinggi dalam data klon memerlukan ukuran sampel yang besar untuk mencapai daya statistik yang memadai. Mendeteksi perbedaan rata-rata 0,3 dalam titer akhir dengan daya 90% akan membutuhkan 141 klon untuk dievaluasi, sementara hanya enam replikasi yang akan cukup dalam populasi kumpulan untuk mencapai hasil yang sama (Gambar 5B ). Meskipun perbedaan ini kurang jelas dalam VCC puncak dan IVCC, mendeteksi efek signifikan masih menuntut replikasi 10–15 kali lebih tinggi dalam klon dibandingkan dengan kumpulan. Persyaratan ini secara dramatis meningkatkan sumber daya, waktu, dan upaya yang diperlukan untuk analisis yang andal. Dengan menggunakan kumpulan, lebih sedikit sampel yang diperlukan, menghasilkan data yang lebih andal dan representatif dan dengan demikian merampingkan proses evaluasi. Selain itu, pendekatan berbasis kumpulan menawarkan penghematan waktu yang substansial, mengurangi jangka waktu untuk menilai KO gen tunggal dari 9 minggu dengan metode klonal menjadi hanya 5 minggu. Efisiensi ini juga memungkinkan peningkatan tiga kali lipat dalam kapasitas penyaringan gen paralel, memungkinkan eksplorasi target genetik yang lebih cepat dan lebih komprehensif. Singkatnya, kumpulan KO tidak hanya menyediakan data yang lebih konsisten dan representatif tetapi juga secara signifikan meningkatkan efisiensi proses rekayasa garis sel. Pendekatan ini memberdayakan kita untuk membuat keputusan yang lebih tepat tentang modifikasi genetik, sambil sangat mengurangi waktu dan sumber daya yang diperlukan untuk penyaringan dan evaluasi.
4 Kesimpulan
Lini sel CHO yang direkayasa secara genetik siap memainkan peran penting dalam masa depan produksi biofarmasi, menawarkan kinerja proses yang ditingkatkan dan kualitas produk yang unggul. Di sini, kami telah mengembangkan jalur yang efisien dan efisien untuk menghasilkan lini sel CHO KO dengan gen KO yang diinginkan. Pendekatan penyaringan gabungan baru kami telah meningkatkan efisiensi evaluasi hingga enam kali lipat, memungkinkan pengiriman cepat lini sel inang dengan produktivitas, karakteristik pertumbuhan, dan kualitas produk yang dioptimalkan. Kemajuan ini membuka jalan bagi peningkatan substansial dalam kinerja proses di seluruh industri biofarmasi, dengan upaya rekayasa yang sedang berlangsung siap untuk lebih memperkuat manfaat ini. Platform penyaringan gabungan memungkinkan pembuatan lini sel CHO KO yang efisien, memfasilitasi studi terperinci tentang karakteristik produksi. Meskipun pendekatan KO gabungan menawarkan alat yang ampuh untuk penyaringan fungsional tahap awal dan korelasi fenotipe, penting untuk dicatat bahwa pembentukan akhir lini sel siap produksi masih memerlukan isolasi dan karakterisasi populasi monoklonal. Misalnya, jika FN1-KO diidentifikasi bermanfaat, lini sel produksi FN1-KO monoklonal harus diturunkan untuk memastikan homogenitas dan stabilitas genetik untuk produksi. Dengan demikian, strategi KO gabungan berfungsi sebagai langkah penyaringan yang cepat dan terukur dalam alur kerja pengembangan lini sel yang lebih luas, yang pada akhirnya mendukung pembuatan lini sel CHO yang lebih aman, lebih hemat biaya, dan lebih berproduksi untuk aplikasi industri.




Tinggalkan Balasan