Ringkasan
- Pada Arabidopsis thaliana , PEMBENTUKAN AKAR LATERAL YANG TERURANG (RLF), protein domain pengikat heme mirip sitokrom b 5 (Cytb5-HBD), diperlukan untuk pembentukan akar lateral (LR) yang tepat. Sementara protein Cytb5-HBD lainnya pada A. thaliana mengatur reaksi metabolisme yang berbeda, RLF bersifat unik karena secara khusus mengatur perkembangan organ. Namun, masih belum diketahui apakah pengikatan heme ke RLF diperlukan untuk fungsinya dan apakah ortolog RLF pada spesies tanaman yang berbeda juga mengatur perkembangan organ.
- Kami menunjukkan bahwa RLF mengikat heme secara in vitro dan bahwa dua residu histidin, yang dilestarikan di antara Cytb5-HBD, sangat penting untuk pengikatan heme dan fungsi biologisnya di A. thaliana . Selain itu, kami menunjukkan bahwa MpRLF, ortolog RLF di bryophyte Marchantia polymorpha , juga mengikat heme secara in vitro dan bahwa Mp RLF menyelamatkan fenotipe pembentukan LR dari mutan rlf A. thaliana .
- Mp rlf ge , mutasi hilangnya fungsi pada Mp RLF , menyebabkan tertundanya pertumbuhan talus dan menghambat pembentukan cawan gemma dan organ reproduksi.
- Temuan kami menunjukkan bahwa MpRLF penting untuk perkembangan vegetatif dan reproduksi yang tepat pada M. polymorpha. Hal ini menunjukkan bahwa sistem reaksi redoks yang bergantung pada RLF dilestarikan di berbagai spesies tanaman dan secara independen digunakan untuk perkembangan organ dalam evolusi tumbuhan lumut dan tumbuhan berbiji.
Perkenalan
Heme adalah kompleks porfirin dengan atom besi yang terkoordinasi secara terpusat. Pada tumbuhan, heme berbagi jalur metabolisme dengan biosintesis Chl hingga produksi protoporfirin IX, setelah itu ferokelatase mengoordinasikan Fe 2+ menjadi protoporfirin IX (Roper & Smith, 1997 ; Hederstedt, 2012 ). Dalam Protein Data Bank ( https://www.rcsb.org ), yang berisi 222.415 protein, 4.272 rantai protein telah diidentifikasi sebagai protein pengikat heme (HBP) dari entri yang berisi tipe heme b dan c (Li et al ., 2011 ). Pengikatan HBP ini ke heme dipertahankan oleh koordinasi asam amino tertentu, seperti histidin, dalam apoprotein ke atom besi heme. Secara umum, dalam berbagai organisme, HBP penting untuk berbagai proses biologis seperti biosintesis steroid, respirasi aerobik, dan kematian sel terprogram karena perannya dalam transfer elektron, oksidasi substrat, dan penyimpanan ion logam (Reedy & Gibney, 2004 ; Layer et al ., 2010 ). Pada tumbuhan, HBP, seperti sitokrom c , yang terlibat dalam transfer elektron selama fotosintesis, dan SOUL4, yang terlibat dalam metabolisme lipid dalam tetesan lemak kloroplas (plastoglobul), telah menunjukkan berbagai efek fisiologis (Kerfeld & Krogmann, 1998 ; Shanmugabalaji et al ., 2020 ). Baru-baru ini, analisis proteomik dalam Arabidopsis thaliana telah mengidentifikasi berbagai HBP, seperti faktor transkripsi nuklir tipe dasar/heliks–loop–heliks dan faktor pensinyalan intraseluler yang terlibat dalam aktivasi GTPase (Shimizu et al ., 2020 ). Penelitian ini telah mengungkap bahwa HBP berfungsi sebagai sinyal intraseluler yang mengatur berbagai fungsi fisiologis. Akan tetapi, masih belum jelas bagaimana HBP terlibat dalam perkembangan organ tanaman.
Satu kelompok HBP pada tumbuhan adalah keluarga protein dengan domain pengikat heme mirip sitokrom b 5 (Cytb5-HBD). Pada tumbuhan, jalur metabolisme yang dimediasi oleh sebagian besar protein Cytb5-HBD beragam, termasuk desaturasi asam lemak, biosintesis lignin, dan reduksi nitrat (Nagano et al ., 2012 ; Gou et al ., 2019 ). Genom A. thaliana memiliki 15 protein dengan Cytb5-HBD, termasuk lima anggota keluarga sitokrom b 5 (At1g26340 (CB5A), At2g32720 (CB5B), At2g46650 (CB5C), At5g48810 (CB5D), dan At5g53560 (CB5E)) (Maggio et al ., 2007 ), satu protein mirip sitokrom b 5 (At1g60660 (CB5LP)), empat protein pengikat progesteron terkait membran (At2g24940 (MAPR2), At3g48890 (MAPR3), At4g14965 (MAPR4), dan At5g52240 (MAPR5)) (Yang et al ., 2005 ), dua Δ-8 sphingolipid desaturase (At3g61580 (SLD1) dan At2g46210 (SLD2)) (Sperling et al ., 1998 ), dua reduktase nitrat (At1g77760 (NR1) dan At1g37130 (NR2)) (Cheng et al ., 1988 ; Wilkinson & Crawford, 1991 ) dan At5g09680/REDUCED LATERAL ROOT FORMATION (RLF) (Ikeyama et al ., 2010 ). Dalam A. thaliana , isoform sitokrom b 5 , SLD1, SLD2, NIA1, NIA2, dan protein dari famili MAPR secara umum tidak memainkan peran signifikan dalam perkembangan organ. CB5D terlibat dalam biosintesis lignin, dan mutan cb5d menunjukkan pengurangan kandungan S-lignin. Namun, tidak ada kelainan dalam perkembangan organ yang diamati pada mutan cb5d (Gou et al ., 2019 ). SLD1 dan SLD2 terlibat dalam desaturasi sphingolipid Δ-8. Mutan yang menyebabkan hilangnya fungsi menunjukkan pertumbuhan akar yang berkurang secara signifikan di bawah tekanan natrium dodecyl sulfate (SDS), tetapi tidak ada cacat pertumbuhan yang diamati dalam kondisi normal (Chen et al ., 2012 ). Demikian pula, NIA1 dan NIA2 terlibat dalam regulasi metabolisme nitrat dan produksi oksida nitrat (NO); namun, mutan null-alel nia1-3 nia2-1 bersifat mematikan, dan tidak ada penelitian yang melaporkan efek pada perkembangan organ. Selain itu, mutan tunggal dari NIA1 atau NIA2 tidak menunjukkan efek signifikan pada pertumbuhan atau perkembangan tanaman (Wilkinson & Crawford, 1993 ; Tang et al ., 2022)). Protein pengikat progesteron yang terkait dengan membran, khususnya MAPR5, berkontribusi terhadap metabolisme lipid dan steroid serta mengatur autofagi dalam kondisi kelaparan, yang berpotensi memengaruhi homeostasis energi. Namun, tidak ada kelainan perkembangan yang parah yang dilaporkan pada mutan MAPR atau galur yang mengalami ekspresi berlebihan (Kimura et al ., 2012 ; Ryu et al ., 2017 ; Wu et al ., 2021 ).
Padahal sebagian besar protein Cytb5-HBD di A. thaliana mengatur berbagai reaksi metabolik, At5g09680/RLF adalah protein Cytb5-HBD unik yang mengatur perkembangan organ, termasuk pembentukan akar lateral (LR) (Ikeyama et al ., 2010 ). Secara umum, pembentukan LR penting untuk arsitektur sistem akar di sebagian besar tumbuhan vaskular dan diatur oleh auksin. Di A. thaliana , pembentukan LR diatur oleh beberapa modul pensinyalan auksin termasuk SOLITARY-ROOT/IAA14, protein auksin/asam indole-3-asetat (Aux/IAA) – AUXIN RESPONSE FACTOR (ARF) 7 dan modul ARF19 (Fukaki et al ., 2005 ; Okushima et al ., 2007 ). Mutan kehilangan-fungsi rlf dari RLF menunjukkan pengurangan yang nyata dalam jumlah LR sementara pensinyalan auksin yang dimediasi ARF7/19 tidak terpengaruh, yang menunjukkan bahwa RLF mengatur pembentukan LR secara independen dari respons auksin (Ikeyama et al ., 2010 ). Selain itu, mutasi rlf menyebabkan perkembangan organ yang tidak normal, termasuk berkurangnya pertumbuhan akar primer dan berkurangnya ukuran daun (Ikeyama et al ., 2010 ). Sementara studi RLF di A. thaliana menjelaskan pentingnya protein Cytb5-HBD untuk pertumbuhan dan perkembangan tanaman, masih ada kesenjangan penting dalam pemahaman kita tentang bagaimana protein Cytb5-HBD mengatur pertumbuhan dan perkembangan organ tanaman. Pertama, tidak diketahui apakah RLF mengikat heme dan bekerja sebagai HBP di planta . Kedua, ada pemahaman yang terbatas tentang apakah protein Cytb5-HBD memiliki peran yang sama dalam mengatur perkembangan organ pada tanaman darat. Untuk mengatasi masalah ini, kami memperluas penyelidikan RLF ke spesies bryophyte model, Marchantia polymorpha (Ishizaki et al ., 2016 ; Fernandez-Pozo et al ., 2022 ) selain A. thaliana . M. polymorpha membentuk organ-organ berbeda yang terspesialisasi untuk fungsi vegetatif atau reproduksi, seperti talus, yang berfungsi sebagai struktur fotosintesis utama, cawan gemma yang menghasilkan gemmae, dan organ reproduksi seperti anteridiofor dan arkegoniofor. Ketertelusuran genetik dan posisi evolusinya sebagai tumbuhan nonvaskular menjadikannya model yang ideal untuk mengeksplorasi pertanyaan-pertanyaan ini.
Dalam penelitian ini, kami menunjukkan bahwa kemampuan pengikatan heme dari RLF diperlukan untuk fungsi biologisnya. Selain itu, kami mengungkap peran Mp RLF , suatu homolog dari RLF dalam M. polymorpha , dalam mengatur perkembangan vegetatif dan reproduksi yang tepat. Temuan kami menyoroti peran penting gen Mp RLF dalam perkembangan organ tanaman. Khususnya, kesamaan antara MpRLF dan A. thaliana RLF menunjukkan adanya mekanisme yang dilestarikan, yang diatur oleh RLF yang secara rumit mengatur perkembangan organ di berbagai garis keturunan tanaman.
Bahan dan Metode
Bahan tanaman dan kondisi pertumbuhan
Aksesi Marchantia polymorpha L. Takaragaike-1 (Tak-1; jantan) dan Takaragaike-2 (Tak-2; betina) digunakan sebagai tipe liar (WT) dalam penelitian ini (Ishizaki et al ., 2008 ). Tanaman Marchantia polymorpha umumnya dikultur pada medium Gamborg’s B5 kekuatan 1/2 yang mengandung agar 1% (b/v) di bawah cahaya kontinyu (50–60 μmol m −2 s −1 ) pada suhu 20°C. Talus divisualisasikan menggunakan mikroskop bedah dan diukur menggunakan perangkat lunak pencitraan Fuji ( Schindelin et al ., 2012 ). Pengamatan cawan gemma dan organ reproduksi dilakukan pada talus M. polymorpha yang dikultur menggunakan mikroskop digital (VHX-5000; Keyence, Osaka, Jepang). Untuk mengamati pertumbuhan rizoid, tanaman dibudidayakan dalam posisi terbalik, dengan media ditempatkan di sisi atas. Pengamatan rizoid dilakukan menggunakan mikroskop stereo M205 FA (Leica Microsystems, Wetzlar, Jerman) yang dilengkapi dengan kamera CCD (DFC7000 T; Leica Microsystems). Untuk mikroskop elektron pemindaian, talus dibekukan dalam nitrogen cair dan diamati dengan mikroskop digital (VHX-D510; Keyence).
Arabidopsis thaliana L. accession Columbia (Col-0) digunakan sebagai WT dalam penelitian ini. Mutasi rlf dan galur transgenik RLFpro:RLF-Green Fluorescent Protein (GFP) telah dideskripsikan sebelumnya (Ikeyama et al ., 2010 ). Benih berkecambah dalam kondisi steril pada media Murashige & Skoog (MS) yang mengandung 1% (b/v) sukrosa yang dipadatkan dengan 0,5% (b/v) gellan gum, seperti yang dideskripsikan sebelumnya (Goh et al ., 2012 ). Tanaman Arabidopsis thaliana ditanam pada suhu 23°C di bawah cahaya kontinyu (40–50 μmol m −2 s −1 ). Jumlah LR dan panjang akar ditentukan menggunakan mikroskop bedah dan perangkat lunak pencitraan Fiji ( Schindelin et al ., 2012 ).
Semua data adalah nilai rata-rata untuk setiap tanaman yang dipertimbangkan. Eksperimen dilakukan tiga kali, dan nilai yang sama diperoleh pada setiap eksperimen. Signifikansi statistik data dihitung menggunakan uji Tukey–Kramer. Analisis statistik pertumbuhan area talus dilakukan menggunakan uji Dunnett, dengan WT sebagai kontrol, dan nilai- P yang disesuaikan dihitung menggunakan metode langkah tunggal. Tingkat signifikansi ditetapkan sebagai berikut: ***, P < 0,001; **, P < 0,01; *, P < 0,05; dan †, P < 0,1.
Konstruksi vektor dan generasi A. thaliana transgenik
Primer yang digunakan untuk konstruksi plasmid tercantum dalam Tabel Informasi Pendukung S1 . Untuk menghasilkan konstruksi RLFpro:RLF-GFP, RLFpro:RLF(H161G)-GFP , RLFpro:RLF(H184G)-GFP , dan RLFpro:RLF(H161G/H184G)-GFP , wilayah promotor RLF ( RLFpro ) (804 bp) diamplifikasi dari DNA genom A. thaliana WT menggunakan primer IF-pGWB501RLFpro-F dan IF-pGWB501RLFpro-R dan dikloning ke dalam pGWB501 menggunakan teknologi In-Fusion (pGWB501 RLFpro ; Takara Bio Inc., Shiga, Jepang). Dengan menggunakan urutan pengkodean yang disintesis sebelumnya (CDS) dari RLF ( RLFcds ) dengan panjang penuh (Ikeyama et al ., 2010 ) sebagai templat, fragmen CACC-RLFcds-XhoI diperkuat menggunakan primer CACC-RLF-F dan RLF-XhoI-R dan dikloning ke dalam vektor kloning pENTR/D-TOPO (Thermo Fisher Scientific, Rockville, MD, AS) untuk menghasilkan vektor pENTR- RLF-XhoI . Fragmen XhoI-GFP-SalI diperoleh dari pBluescript II SK(+) GFP melalui perlakuan enzim restriksi dan diligasikan dengan pENTR -RLF-XhoI yang dibelah oleh enzim restriksi untuk menghasilkan pENTR- RLF-GFP . Fragmen pENTR -RLF(H161)-GFP dan pENTR- RLF(H184)-GFP dibuat dengan menggunakan PrimeSTAR Max DNA Polymerase (Takara Bio Inc.) dan primer H161G-F/H161G-R dan H184G-F/H184G-R. Selain itu, fragmen pENTR- RLF(H161G/H184G)-GFP dibuat dengan cara yang sama menggunakan primer H184G-F/H184G-R dengan pENTR- RLF(H161)-GFP sebagai templat. Vektor entri ini dan pGWB501- RLFpro diperlakukan dengan campuran enzim Gateway LR klonase II (Thermo Fisher Scientific) untuk menghasilkan konstruksi RLFpro:RLF-GFP , RLFpro:RLF(H161G)-GFP , RLFpro:RLF(H184G)-GFP , dan RLFpro:RLF(H161G/H184G)-GFP . Untuk menghasilkan RLFpro: Mp RLF , vektor pGWB501- RLFpro dan pENTR-Mp RLFcds (akan dijelaskan kemudian) direaksikan dengan campuran enzim Gateway LR klonase II (Thermo Fisher Scientific). Vektor biner ini ditransformasikan menjadi mutan rlf-1 menggunakan metode celupan bunga (Clough & Bent, 1998 ) dengan Agrobacterium tumefaciens (galur C58MP90).
Konstruksi vektor dan generasi M. polymorpha transgenik
Primer yang digunakan untuk konstruksi plasmid tercantum dalam Tabel S1 . Untuk menghasilkan konstruksi Mp4g04390/ Mp RLFpro:GUS , wilayah promoter Mp RLF (Mp RLFpro ) diamplifikasi melalui PCR dari DNA genom Tak-1 sebagai urutan sepanjang 4411 bp yang mengandung kodon awal pada ekson pertama menggunakan primer MpRLFpro-CACC-F dan MpRLFpro-R. Fragmen yang diamplifikasi dikloning ke dalam vektor kloning pENTR/D-TOPO (Thermo Fisher Scientific) untuk menghasilkan vektor pENTR-Mp RLFpro . Wilayah Mp RLFpro dari vektor pENTR-Mp RLFpro dimasukkan ke dalam vektor biner Gateway pMpGWB104 (Ishizaki et al ., 2015 ) menggunakan campuran Enzim Gateway LR clonase II (Thermo Fisher Scientific) untuk menghasilkan konstruksi biner Mp RLFpro:GUS . Vektor Mp RLFpro:GUS dimasukkan ke dalam talus regenerasi Tak-1 menggunakan A. tumefaciens GV2260 seperti yang dijelaskan sebelumnya (Kubota et al ., 2013 ). Transforman dipilih menggunakan 100 μg ml −1 higromisin B dan 100 μg ml −1 sefotaksim. Mutasi loss-of-function dari Mp RLF (Mp rlf ge ) dihasilkan menggunakan sistem CRISPR/Cas9 seperti yang dijelaskan sebelumnya (Sugano et al ., 2018 ; Sugano & Nishihama, 2018 ). Kami memilih urutan target pada persimpangan intron kedua dan ekson ketiga dari Mp RLF . Oligonukleotida sintetis untuk situs target yang ditunjukkan pada Tabel S1 dianneal, dimasukkan ke dalam pMpGE_En03 yang dicerna BsaI menggunakan T4 DNA Ligase (Takara Bio Inc.), dan kemudian dimasukkan ke dalam vektor tujuan pMpGE010 atau pMpGE011 menggunakan campuran Enzim Gateway LR clonase II (Sugano et al ., 2018 ; Sugano & Nishihama, 2018 ). Vektor diperkenalkan ke dalam regenerasi thalli Tak-1 atau gemmae Tak-2 menggunakan Agrobacterium tumefaciens GV2260 (Kubota et al ., 2013 ; Tsuboyama et al ., 2018 ), dan transforman dipilih menggunakan 100 μg ml −1 hygromycin atau 0,5 μM chlorsulfuron. Untuk komplementasi Mp rlf ge , cds dari Mp RLF dengan panjang penuh diperkuat dengan reaksi berantai polimerase transkriptase balik menggunakan KOD-Plus-Neo (TOYOBO, Osaka, Jepang) dengan primer MpRLFcds-CACC-F dan MpRLFcds (+stop)-R. Fragmen urutan pengkodean Mp RLF dikloning ke dalam vektor kloning pENTR/D-TOPO (Thermo Fisher Scientific) untuk menghasilkan pENTR-Mp RLFcds . Untuk mencegah CRISPR-Cas9 menargetkan Mp RLFcds , substitusi urutan sinonim diperkenalkan antara posisi nukleotida 472 dan 492 (asam amino dari G158 hingga R164) dalam Mp RLFcds sebagai berikut. pENTR-Mp RLFcds digunakan sebagai cetakan, dan seluruh plasmid diamplifikasi menggunakan PrimeSTAR Max DNA Polymerase (Takara Bio Inc.) dan primer IF-MpRLF(com)-F dan IF-MpRLF(com)-R untuk menghasilkan pENTR-Mp RLFcds_resistant , Mp RLFcds yang tahan CRISPR-Cas9 . Wilayah promoter Mp RLF (Mp RLFpro ), termasuk 4408-bp di hulu kodon inisiasi, diamplifikasi dari DNA genom Tak-1 melalui PCR menggunakan PrimeSTAR Max (Takara Bio Inc.) dengan primer IF-MpRLFpro-F dan IF-MpRLFpro-R. Produk yang diamplifikasi dengan PCR dikloning ke dalam situs HindIII dari pMpGWB301 (Ishizaki et al ., 2015 ) menggunakan kit kloning In-Fusion HD (Clontech, Mountain View, CA, AS). Urutan pengkodean MpRLF yang resistan terhadap CRISPR-Cas9 dalam pENTR-Mp RLFcds_resistant dimasukkan ke dalam vektor biner menggunakan campuran Enzim Gateway LR clonase II (Thermo Fisher Scientific) untuk menghasilkan konstruksi Mp RLFpro: Mp RLFcds_resistant . Vektor Mp RLFpro: Mp RLFcds_resistant dimasukkan ke dalam gemmae mutan Mp rlf ge menggunakan A. tumefaciens GV2260. Metode terperinci dijelaskan dalam penelitian sebelumnya (Tsuboyama et al ., 2018 ). Untuk menghasilkan Mp E2Fpro:XVE>> Mp RLFcds , urutan pengkodean Mp RLF diperkuat dengan PCR menggunakan PrimeSTAR Max (Takara Bio Inc.) dengan set primer MpRLFcds-CACC-F dan MpRLFcds(−stop)-R dan dikloning ke dalam vektor kloning pENTR/D-TOPO. Reaksi akar lateral dilakukan pada sisi terminal-C dari vektor tujuan fusi GFP berdasarkan vektor biner Gateway pMpGWB168 (Ishida et al ., 2022 ) dan vektor entri. Vektor Mp E2Fpro:XVE>> Mp RLFcds-GFP dimasukkan ke dalam talus regenerasi Tak-1 seperti yang dijelaskan sebelumnya (Kubota et al ., 2013 ). Transforman dipilih dengan 100 μg ml −1 higromisin B dan 100 μg ml −1 sefotaksim. Untuk mengevaluasi efek dari ekspresi berlebih bersyarat dari Mp RLF , gemmae dari tanaman Mp EFpro:XVE>> Mp RLF-GFP dikultur selama 7 hari pada pelat medium Gamborg’s B5 kekuatan 1/2, diikuti oleh 7 hari pada pelat medium Gamborg’s B5 kekuatan 1/2 yang mengandung 10 μM β-estradiol (EST; FUJIFILM Wako Pure Chemical Corp., Osaka, Jepang).
Untuk menghasilkan konstruksi Mp RLFpro:RLFcds , urutan pengkodean panjang penuh RLF ( RLFcds ) diperkuat menggunakan PrimeSTAR Max DNA Polymerase (Takara Bio Inc.) dengan set primer CACC-RLF-F dan RLF-stop-R. RLFcds yang disintesis sebelumnya digunakan sebagai cetakan untuk amplifikasi (Ikeyama et al ., 2010 ). Fragmen RLFcds yang diperkuat dimasukkan ke dalam vektor pENTR/D-TOPO (Thermo Fisher Scientific) melalui reaksi TOPO untuk membuat RLFcds/pENTR . Menggunakan RLFcds/pENTR dan vektor biner pMpGWB301 yang mengandung wilayah promotor Mp RLF (Mp RLFpro ), konstruksi Mp RLFpro:RLFcds dihasilkan dengan Gateway LR Clonase II Enzyme Mix (Thermo Fisher Scientific). Vektor Mp RLFpro:RLFcds ditransformasikan ke dalam gemmae mutan Mp rlf ge menggunakan A. tumefaciens GV2260. Metode terperinci dijelaskan dalam penelitian sebelumnya (Tsuboyama et al ., 2018 ).
Pewarnaan GUS
Untuk uji aktivitas β-Glucuronidase (GUS) histologis, talus berumur 14 hari dan organ reproduksi (diinduksi oleh cahaya putih selama 3 minggu diikuti oleh cahaya merah jauh dan cahaya putih selama 7 minggu) dari Mp RLFpro:GUS /Tak-1 dihilangkan udaranya dua kali selama 5 menit, diinkubasi dalam larutan pewarnaan GUS (0,5 mM kalium ferrosianida, 0,5 mM kalium ferrisianida, dan 1 mM X-Gluc) pada suhu 37°C selama 4 jam. Sampel kemudian dibersihkan dengan etanol 70% (v/v). Gemmae diinkubasi dalam larutan pewarnaan GUS yang sama selama 24 jam dalam kondisi yang sama. Sampel talus yang diwarnai dengan β-Glucuronidase ditanamkan dalam agar 6% (b/v). Potongan sampel berukuran 100–130 μm dipotong menggunakan MicroSlicer (DOSAKA DTK-1000; Dosaka EM Co., Ltd, Kyoto, Jepang). Arkegoniofor yang diwarnai dengan β-Glukoronidase dikeringkan dan ditanamkan dalam resin Technovit 7100 (Heraeus-Kulwer, Wehrheim, Jerman) sesuai dengan protokol pabrik pembuatnya. Potongan tipis (5 μm) dipotong menggunakan mikrotom (HM325; MICROM International Inc., Walldorf, Jerman), dikeringkan, dan diwarnai ulang untuk dinding sel dengan 0,05% ruthenium merah (Nacalai Tesque Inc., Kyoto, Jepang). Potongan tersebut dicitrakan dengan mikroskopi kontras interferensi diferensial (mikroskop Leica DM6000; Leica Microsystems) dan dirakit oleh e-Tilling (Mitani Shoji Co., Ltd., Tokyo, Jepang).
Mikroskopi pemindaian laser konfokal
Gemmae yang mengekspresikan MpRLF-GFP yang dapat diinduksi EST diwarnai tandingan dengan 10 μg ml −1 propidium iodida (PI) selama 5 menit dan diamati dengan mikroskop pemindaian laser confocal menggunakan mikroskop confocal FV1000 (Olympus/Evident, Tokyo, Jepang). Garis 473-nm dan garis 559-nm dari laser LD digunakan untuk merangsang GFP dan PI, masing-masing. Gambar diperoleh menggunakan lensa objektif UPlanSAPO 40x/0,95 (Olympus/Evident). Gambar diproses menggunakan program Fiji / Image J (Schindelin et al ., 2012 ).
Penyelarasan sekuens ganda dan analisis filogenetik
Urutan asam amino A. thaliana RLF (At5g09680) digunakan sebagai templat untuk mengidentifikasi ortolog RLF pada spesies tanaman lain menggunakan PSI-B last NCBI (Position-Specific Iterative B last ; https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=psiblast ; Altschul et al ., 1997 ), B last P dari P hytozome ( https://phytozome-next.jgi.doe.gov/ ; Goodstein et al ., 2012 ), dan data transkriptomik dari 1000 Plant Transcriptomes Project ( https://db.cngb.org/onekp/ ; Leebens-Mack et al ., 2019 ). Ortolog RLF yang diidentifikasi pada setiap spesies tumbuhan dan alga tercantum dalam Tabel 1 , dan sekuensnya dikenakan penyelarasan sekuens ganda menggunakan M afft . Selain itu, sekuens asam amino A. thaliana CB5-D (At5g48810) digunakan sebagai templat untuk mengidentifikasi sekuens protein CB5 yang serupa (CB5A, B, C, D, E, dan LP) pada spesies tumbuhan lain. Sekuens ortolog RLF dan CB5 dari setiap tumbuhan dipilih berdasarkan ambang batas nilai-E 1e-15. Penyelarasan sekuens ganda dari sekuens asam amino yang diperoleh dilakukan menggunakan C lustal W (Thompson et al ., 1994 ; Waterhouse et al ., 2009 ; Katoh & Standley, 2013 ). Selanjutnya, pohon filogenetik tanpa akar dibangun berdasarkan urutan asam amino CytB5 HBD dari protein RLF dan CB5 (CB5A, B, C, D, E, dan LP) dari A. thaliana (At5g09680), Glycine max (Wm82.a6 Glyma.12G009100 dan Glyma.09G227600), Oryza sativa (Os07g0232200), Amborella trichopoda (AmTr_v1.0_scaffold00029.68), Thuja plicata (Thupl.29380466s0005), Selaginella moellendorffii (127 066 dan 138 045), Marchantia polymorpha (Mp3g04390), Physcomitrium patens (Pp6c20_12240V6), Phaeoceros carolinianus (RXRQ_scaffold_2017981), Chlamydomonas reinhardtii (Cre13.g574800), Volvox carteri (Voccar.0001s0218), Chara braunii (CBR_g29320), dan Porphyra umbilicalis (Pum0348s0012). Setiap klade dalam pohon filogenetik diberi label sesuai dengan klasifikasinya (misalnya klade RLF, klade CB5A/C). Pohon tersebut dibangun menggunakan metode kemungkinan maksimum (1000 uji bootstrap), model WAG (Whelan dan Goldman) (Whelan & Goldman, 2001), dan perangkat lunak Mega 11 (Tamura et al ., 2021 ). Nilai bootstrap (1000 pengujian) ditampilkan sebagai simpul berkode warna, yang menunjukkan tingkat keyakinan masing-masing. Pohon filogenetik molekuler dibangun menggunakan tvBOT (Xie et al ., 2023 ). Jarak evolusi ditampilkan di bawah cabang.
| Jenis | Versi | Kelompok | ortolog RLF |
|---|---|---|---|
| Arabidopsis thaliana | TAIR10 (Sumber Informasi Arabidopsis) | Tumbuhan Berbiji tertutup | 1 |
| Glisin maks | Wm82.a6.v1 ( P.hitozoa ) | Tumbuhan Berbiji tertutup | 2 |
| Tanaman Oryza sativa | v.7.0 ( P.hytozoa ) | Tumbuhan Berbiji tertutup | 1 |
| Jamur Amborella trichopoda | v.1.0 ( P.Hitozoa ) | Tumbuhan Berbiji tertutup | 1 |
| Thuja plicata | v.3.1 ( P.Hitozoa ) | Tumbuhan Gymnospermae | 1 |
| Selaginella moellendorffii | v.1.0 ( P.Hitozoa ) | Likofit | 2 |
| Marchantia polimorfa | v.7.1 (MarpolBase) | Lumut hati (Bryophyta) | 1 |
| Physcomitrella patens | v.6.1 ( P.Hitozoa ) | Lumut (Bryophyta) | 1 |
| Burung pipit | RXRQ (1000KP) | Lumut tanduk (Bryophyta) | 1 |
| Chlamydomonas reinhardtii | CC-4532 v6.1 (P. hytozoa ) | Ganggang Hijau | 1 |
| Volvox carter | v.2.1 ( P.Hitozoa ) | Ganggang Hijau | 1 |
| Chara coklat | Cbr_1.0 (NCBI) | Karofit | 1 |
| Porphyra umbilicalis | v.1.5 ( P.Hitozoa ) | Ganggang Merah | 1 |
Setiap baris menyertakan nama spesies, versi, klasifikasi kelompok, dan jumlah ortolog RLF yang sesuai.
Kesamaan urutan asam amino antara RLF dan MpRLF dievaluasi menggunakan EMBOSS Matcher untuk urutan panjang penuh, wilayah N-terminal, dan CytB5 HBD ( https://www.ebi.ac.uk/jdispatcher/psa/emboss_matcher ; Madeira et al ., 2024 ).
Pemurnian protein dan pengukuran spektrum absorbansi
Pemurnian protein rekombinan dilakukan menurut protokol Sistem Pemurnian dan Fusi Protein pMAL (New England Biolabs, Ipswich, MA, AS). Klon ekspresi maltose-binding protein (MBP)-RLF diperoleh melalui reaksi LR dari Vektor pMal-c5X, yang mengandung urutan att R1, gen ccdB , dan urutan att R2 pada situs multikloning, dengan RLFcds/pENTR. Untuk menghasilkan RLF mutan MBP, kami menghasilkan konstruksi RLFcds(H161A) /pENTR, RLFcd(H184A) /pENTR dengan menggunakan RLFcds /pENTR sebagai templat dengan primer H161A-F/H161A-R dan H184A-F/H184-R menggunakan PrimeSTAR Max DNA Polymerase (Takara Bio Inc.). Untuk konstruksi RLFcds(H161A/H184A)/ pENTR, RLFcds(H161A) /pENTR digunakan sebagai templat menggunakan primer H184A/-F/H184-R dengan PrimeSTAR Max DNA Polymerase (Takara Bio Inc.). Mutasi RLF/ pENTR ini digunakan dengan cara yang sama untuk melakukan reaksi LR guna membangun MBP-RLF(H161A), MBP-RLF(H184A) , dan MBP-RLF(H161A.H184A) . Demikian pula, klon ekspresi untuk MBP-MpRLF juga dibangun menggunakan metode yang sama melalui reaksi LR dengan Mp RLFcds /pENTR. Klon ekspresi ditransformasikan ke dalam E. coli JM109. Koloni yang tumbuh dikulturkan terlebih dahulu dalam medium cair LB 5 ml yang mengandung 50 μg ml −1 ampisilin dan 0,2% (b/v) glukosa pada suhu 37°C selama 18 jam. Dari medium prakultur, 3 ml ditambahkan ke 300 ml medium cair LB yang mengandung 50 μg ml −1 ampisilin dan 0,2% (b/v) glukosa pada suhu 37°C, 1,31 g selama 2–3 jam inkubasi, diikuti dengan pengocokan pada suhu 20°C, 1,31 g selama 1 jam. Dari 100 mM isopropil β-D-tioglaktopiranosida (Takara Bio Inc.), 1 ml ditambahkan ke medium kultur, dan campuran dikocok pada suhu 20°C, 0,73 g selama 20 jam. Pelet sel dikumpulkan dengan sentrifugasi pada kecepatan 1175 g selama 10 menit pada suhu 4°C, disuspensikan kembali dalam 1× PBS, dan disentrifugasi lagi pada kecepatan 1175 g selama 10 menit pada suhu 4°C. Pelet sel yang terkumpul disimpan pada suhu -80°C.
Setelah digunakan, pelet disuspensikan dalam 5 ml Buffer A (20 mM Tris–HCl pH 7,5, 200 mM NaCl) dan dipindahkan ke gelas kimia 10 ml. Untuk menekan aktivitas enzim proteolitik, 20 μl 10 mg ml −1 fenilmetilsulfonil fluorida (PMSF; Sigma-Aldrich, St. Louis, MO, AS) ditambahkan ke suspensi bakteri sebelum lisis dengan sonikasi. Setelah perlakuan lisis, ekstrak dialiquot ke dalam tabung 1,5 ml, disentrifugasi pada suhu 4°C, 16.000 g selama 15 menit, dan supernatan dikumpulkan dalam tabung 50 ml. Dari resin amilosa (bubur 50% (b/v), ditambahkan 300 μl dan didiamkan pada suhu 4°C selama 30 menit dengan pengadukan sedang. Campuran tersebut kemudian disentrifugasi pada suhu 4°C selama 1 menit pada 500 g , dan supernatan dibuang. Selanjutnya, 1 ml Buffer A ditambahkan ke endapan resin dan dikumpulkan dalam tabung 2 ml. Setelah dicuci, 250 μl Buffer B (20 mM Tris–HCl pH 9,5, 200 mM NaCl, dan 10 mM maltosa) ditambahkan ke resin, dengan supernatan dibuang, dan didiamkan pada suhu 4°C selama 30 menit dengan pencampuran sedang. Setelah disentrifugasi pada suhu 4°C, 500 g selama 1 menit, supernatan dikumpulkan, dan 6 × Buffer stok (60% (v/v) gliserol, 0,3% (v/v) Triton-100) ditambahkan sebelum disimpan pada suhu −80°C.
Untuk mengukur spektrum serapan ultraviolet (UV)-tampak protein RLF, sampel disentrifugasi untuk menghilangkan pengotor dan dipertukarkan dengan ultrafiltrasi ke dalam buffer yang mengandung 20 mM Tris–HCl (pH 8,0), 500 mM NaCl, dan 6% (v/v) gliserol. Konsentrasi protein disesuaikan menjadi 1,5 μM. Kelebihan natrium ferisianida dalam larutan dan beberapa butir ditionit padat ditambahkan ke sampel untuk masing-masing menetapkan kondisi heme teroksidasi dan tereduksi. Pengukuran spektrum serapan dilakukan menggunakan spektrofotometer UV-1900i (Shimadzu, Kyoto, Jepang).
RNA-seq dan analisis transkriptomik
Total RNA dimurnikan dari talus berumur 16 hari ( sekitar 100 mg berat segar) menggunakan RNeasy Plant Mini Kit (Qiagen, Hilden, Jerman); RNA diukur menggunakan spektrofotometer NanoDrop ND1000 (Thermo Fisher Scientific). Alikuot RNA sebanyak 10 μg ml −1 atau lebih dari tiga replikasi biologis digunakan untuk analisis RNA-Seq. Persiapan pustaka dan analisis RNA-seq dikontrakkan ke BGI Jepang ( https://www.bgi.com/jp/home ), dan pembacaan bersih diperoleh dari setiap pustaka. Hasil pembacaan dipetakan menggunakan MpTak_v6.1r2 dari Marpolbase ( https://marchantia.info/ ), dengan tidak memasukkan sekuens pada kromosom U dari data. Kami menggunakan Hisat 2 (Kim et al ., 2015 ) untuk menyelaraskan pembacaan bersih dengan genom referensi dan kemudian String Tie ( Pertea et al ., 2015 ) untuk merakitnya. Akhirnya, hasil perakitan semua sampel digabungkan menggunakan Cuffmerge . Kami menggunakan Bowtie 2 (Langmead & Salzberg, 2012 ) untuk menyelaraskan pembacaan bersih dengan urutan referensi, dan kemudian menggunakan RSEM (Li & Dewey, 2011 ) untuk menghitung ekspresi gen dan transkrip. Analisis pengayaan menggunakan Gene Ontology (GO) dan Kyoto Encyclopedia of Genes and Genomes (KEGG) untuk mengklasifikasikan fungsi gen dan mengevaluasi kelimpahan gen dalam modul fungsional tertentu. Secara statistik, model hipergeometrik menghitung nilai – P untuk setiap modul fungsional. Koreksi tingkat penemuan palsu (FDR) dilakukan untuk menentukan pengayaan yang signifikan; modul fungsional dengan FDR 0,01 atau kurang dianggap signifikan. Bagan gelembung dan tampilan hierarki GO didasarkan pada hasil analisis pengayaan GO menggunakan G oatools (Klopfenstein et al ., 2018 ). Setiap gelembung menunjukkan jumlah gen yang terkait dengan istilah GO, dan warna mewakili skor pengayaan (−log 10 (p_fdr_bh)). −log 10 (p_fdr_bh) adalah nilai yang dihasilkan dari transformasi logaritmik nilai p_fdr_bh dengan basis 10 dan inversi tanda. Nilai p_fdr_bh dihitung dengan mempertimbangkan beberapa perbandingan menggunakan metode Benjamin–Hochberg. Metode ini diterapkan pada p_uncorrected, yang menunjukkan apakah setiap istilah GO diperkaya secara signifikan. Tampilan hierarki GO memvisualisasikan hubungan antara setiap istilah GO menggunakan Graphviz pada hasil analisis pengayaan GO (Ellson et al ., 2002 ; Klopfenstein et al ., 2018)). Setiap istilah GO mencakup nilai p_uncorrected yang ditransformasikan ke dalam logaritma. Semua data RNA-Seq untuk Tak-1 dan mutan Mp rlf ge disimpan dalam NCBI Sequence Read Archive (SRA) (PRJNA1225401).
Hasil
Kemampuan mengikat heme diperlukan untuk fungsi RLF di A. thaliana
Dua residu histidin dalam Cytb5-HBD diketahui penting untuk pengikatan heme (Cowley et al ., 2002 ; Schenkman & Jansson, 2003 ). Perbandingan urutan asam amino Cytb5-HBD dalam RLF dengan protein Cytb5-HBD lainnya dalam A. thaliana mengungkapkan bahwa dua residu histidin pada posisi 161 dan 184 dari RLF sesuai dengan situs pengikatan heme yang diprediksi dan dilestarikan di antara semua protein Cytb5-HBD kecuali untuk keluarga MAPR (Gbr. 1a , S1a ). Untuk mengonfirmasi pentingnya residu histidin ini, kami memurnikan protein WT rekombinan dan RLF mutan dengan substitusi histidin-ke-alanin individual atau gabungan (H161A, H184A, dan H161A/H184A; Gbr. S1b ) dan mengukur spektrum absorbansinya. Pelet sel E. coli yang mengekspresikan WT MBP-RLF tampak berwarna cokelat kemerahan, yang menunjukkan keberadaan heme, sedangkan pewarnaan ini tidak teramati pada RLF mutan (Gbr. S1c ). Analisis elektroforesis gel poliakrilamid–natrium dodecyl sulfate (SDS-PAGE) mengonfirmasikan ekspresi dan pemurnian MBP-RLF dan MBP-RLF mutan (Gbr. S1d ). Bentuk teroksidasi dari WT RLF menunjukkan puncak pada pita soret (412 nm) yang merupakan karakteristik protein Cytb5-HBD. Sebaliknya, bentuk tereduksi dari RLF menunjukkan puncak pada pita αβ (527 dan 557 nm) selain pita soret (423 nm) (Gbr. 1b ). Spektrum serapan WT RLF ini merupakan karakteristik protein Cytb5-HBD (Smith et al ., 1994 ; Wahl et al ., 2010 ). Sebaliknya, spektrum protein RLF mutan (H161A, H184A, dan H161A/H184A) menunjukkan tidak adanya puncak karakteristik ini, yang mengonfirmasi peran penting residu histidin dalam pengikatan heme (Gbr. 1b ). Struktur setiap protein RLF mutan, yang diprediksi menggunakan Colab Fold ( Mirdita et al ., 2022 ), menunjukkan bahwa CytB5-HBD C-terminal sangat terkonservasi antara protein WT dan mutan (Gbr. S1b ). Hal ini menunjukkan bahwa substitusi situs pengikatan heme tidak memengaruhi keseluruhan struktur protein . Hasil ini mengindikasikan bahwa RLF adalah HBP dan mengonfirmasi bahwa dua residu histidin pada posisi 161 dan 184 diperlukan untuk pengikatan heme ke RLF secara in vitro .
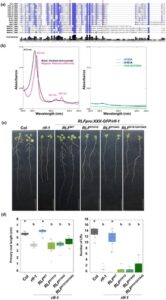
Meskipun RLF mengikat heme secara in vitro , peran fungsional pengikatan heme ke RLF di planta masih belum jelas. Untuk menjawab pertanyaan ini, kami menghasilkan tanaman A. thaliana transgenik yang mengekspresikan RLF mutan di mana residu histidin pada posisi 161 dan 184, secara individual atau dalam kombinasi, digantikan dengan glisin (H161G, H184G, dan H161G/H184G), dalam latar belakang mutan rlf-1 (Ikeyama et al ., 2010 ). Seperti yang ditunjukkan sebelumnya, mutan rlf-1 menunjukkan pengurangan yang nyata dalam jumlah LR dan mengurangi pertumbuhan akar primer, dibandingkan dengan WT (Gbr. 1c,d ). Transgen RLFpro:RLF-GFP yang mengekspresikan RLF WT berlabel GFP berhasil menyelamatkan fenotipe mutan rlf-1 (Gbr. 1c,d ). Sebaliknya, ekspresi mutan RLF berlabel GFP ( RLFpro:RLF(H161G)-GFP, RLFpro:RLF(H184G)-GFP , dan RLFpro:RLF(H161G/H184G)-GFP ) di bawah kendali promotor RLF gagal memulihkan pembentukan LR dan pertumbuhan akar primer pada mutan rlf-1 (Gbr. 1c,d ). Hasil ini menunjukkan bahwa dua residu histidin pada posisi 161 dan 184 penting untuk fungsi RLF pada tanaman , yang menyiratkan bahwa pengikatan heme diperlukan untuk fungsi biologis RLF.
Konservasi RLF pada berbagai spesies tanaman
Penelitian sebelumnya telah menunjukkan bahwa gen ortolog RLF terdapat pada beberapa tanaman, termasuk padi ( Oryza sativa ), monokotil, dan lumut Physcomitrium patens (Ikeyama et al ., 2010 ). Bahasa Indonesia: Untuk mengonfirmasi konservasi urutan asam amino di antara ortolog RLF, kami pertama-tama melakukan beberapa perbandingan penyelarasan menggunakan J alview (Waterhouse et al ., 2009 ) pada urutan asam amino dari A. thaliana (At5g09680), Glycine max (Wm82.a6 Glyma.12G009100, Wm82.a6 Glyma.09G227600), Oryza sativa ( LOC_Os07g12830 ), Amborella trichopoda (AmTr_v1.0_scaffold00029.68), Thuja plicata ( Thupl.29380466s0005 ), Selaginella moellendorffii (127 066, 138 045), M. polymorpha (Mp3g04390), Physcomitrium patens (Pp3c20_23480), Phaeoceros carolinianus (RXRQ_scaffold_2017981), Chlamydomonas reinhardtii (Cre13_g 574 800), Volvox carteri (Vocar.0001s0218), Chara braunii (CBR_g29320), dan Porphyra umbilicalis (Pum0348s0012). Analisis ini mengungkapkan bahwa daerah Cytb5-HBD C-terminal sangat terkonservasi di antara ortolog RLF (Gbr. S2a ), sementara panjang dan konservasi daerah yang tidak teratur pada sisi N-terminal bervariasi secara signifikan di antara ortolog ini (Gbr. S2a ). Khususnya, dua residu histidin, yang merupakan bagian dari situs pengikat heme, sangat terpelihara di bagian terminal C dari wilayah CytB5-HBD (Gbr. S2b ). Temuan ini menunjukkan bahwa pengikatan heme sangat penting bagi fungsi ortolog RLF.
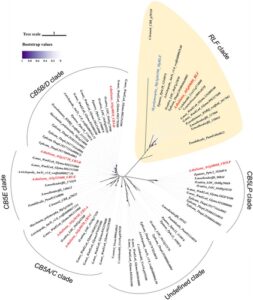
Kami selanjutnya menyusun pohon filogenetik molekuler yang mencakup baik ortolog RLF maupun anggota famili CB5 untuk memeriksa konservasi evolusioner mereka di seluruh kingdom tumbuhan (Gbr. 2 ). Analisis ini mengungkapkan bahwa ortolog RLF membentuk klade yang berbeda dan independen yang dilestarikan di berbagai tumbuhan, termasuk angiospermae, gymnospermae, likofit, lumut hati, lumut kerak, alga karofit, dan klorofit (Tabel 1 ). Pohon filogenetik ini konsisten dengan penelitian sebelumnya tentang filogeni CB5, seperti pengelompokan CB5B dan CB5D dalam klade yang sama, yang mendukung validitas pohon tersebut (Zhao et al ., 2024 ). Lebih jauh lagi, sebagian besar spesies tumbuhan memiliki satu ortolog RLF , kecuali Glycine max dan Selaginella moellendorffii (Tabel 1 ). Temuan-temuan ini menunjukkan bahwa pohon filogenetik molekuler selaras dengan klasifikasi yang ditetapkan sebelumnya sambil dengan jelas membedakan keunikan evolusi klade RLF.
Mp RLF , homolog RLF di M. polymorpha , melengkapi fenotipe mutan rlf di A. thaliana
Meskipun ortolog RLF dilestarikan di antara tumbuhan, kami tidak tahu apakah mekanisme yang bergantung pada RLF dalam perkembangan organ juga dilestarikan di seluruh spesies. Untuk memeriksa apakah ortolog RLF ini juga mengatur perkembangan organ, kami menggunakan spesies lumut model M. polymorpha dan menamai ortolog RLF M. polymorpha Mp RLF . Kami melakukan analisis penyelarasan menggunakan EMBOSS Matcher ( https://www.ebi.ac.uk/jdispatcher/psa/emboss_matcher ; Madeira et al ., 2024 ) dan prediksi struktural menggunakan A lpha Fold 2 (dimodifikasi dari uniport; https://www.uniprot.org ; Skolnick et al ., 2021 ). Hal ini menunjukkan bahwa MpRLF dan Arabidopsis RLF memiliki 51,3% kesamaan sekuens keseluruhan, dengan konservasi yang sangat tinggi pada domain Cytb5-HBD (86,3%), yang penting untuk aktivitas pengikatan heme (Gbr. 3a ). Mengingat kesamaan tersebut, kami menguji apakah MpRLF menunjukkan spektrum pengikatan heme yang mirip dengan Cytb5-HBD dengan melakukan uji pengikatan heme menggunakan protein MpRLF rekombinan. Mirip dengan Arabidopsis RLF, pelet sel E. coli yang mengekspresikan WT MBP-MpRLF tampak berwarna coklat kemerahan, yang menunjukkan adanya heme, dan analisis SDS-PAGE mengonfirmasi ekspresi dan pemurnian MBP-MpRLF (Gbr. S1c,d ). Protein MpRLF WT menunjukkan spektrum pengikatan heme yang khas, konsisten dengan yang diamati untuk A. thaliana RLF (Gbr. 3b ). Hasil-hasil ini menegaskan bahwa MpRLF memiliki aktivitas pengikatan heme yang terpelihara, mendukung hipotesis bahwa sifat ini terpelihara di seluruh RLF tumbuhan darat.
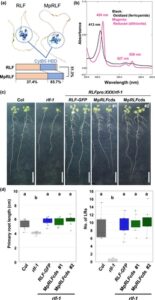
Untuk menilai apakah fungsi gen Mp RLF dilestarikan antara M. polymorpha dan A. thaliana , kami mengekspresikan Mp RLFcds di bawah kendali promotor RLF dalam latar belakang mutan rlf A. thaliana ( RLFpro: Mp RLFcds/rlf-1 ). Seperti yang ditunjukkan pada Gambar 3 , ekspresi MpRLF dalam mutan rlf sepenuhnya memulihkan pengurangan jumlah LR dan fenotipe panjang akar primer (Gambar 3c,d ). Hasil ini menunjukkan bahwa MpRLF secara fungsional dapat dipertukarkan dengan Arabidopsis RLF, yang menunjukkan bahwa mekanisme yang bergantung pada RLF dalam perkembangan organ dilestarikan dan telah diadopsi secara independen di A. thaliana dan M. polymorpha .
Mp RLF diekspresikan sepanjang pelepah talus di M. polymorpha
Berikutnya, kami meneliti pola ekspresi Mp RLF di M. polymorpha . Basis data M. polymorpha yang baru-baru ini diterbitkan , Marpolbase Expression ( https://marchantia.info/mbex/ ; Kawamura et al ., 2022 ), menunjukkan bahwa Mp RLF diekspresikan secara tinggi pada arkegoniofor dan arkegonium dewasa (Gbr. S3 ). Ekspresi Mp RLF juga diamati pada anteridiofor (yang menopang dan mengangkat anteridia), talus, cawan gemma, dan pelepah daun. Sebaliknya, tingkat ekspresi sangat rendah pada sel apikal gametofit dan sporofit muda (Gbr. S3 ). Hasil ini menunjukkan bahwa ekspresi Mp RLF dikaitkan dengan perkembangan organ selama fase pertumbuhan vegetatif dan reproduksi. Untuk menjelaskan pola ekspresi Mp RLF secara terperinci, kami menghasilkan tanaman M. polymorpha transgenik yang mengekspresikan gen reporter GUS di bawah kendali promotor Mp RLF (Mp RLFpro:GUS ). Dalam garis Mp RLFpro:GUS , aktivitas GUS dideteksi melalui pewarnaan histologis di daerah tengah gemmae dan di pelepah daun dan dekat meristem apikal talus berumur 14 hari. Sebaliknya, aktivitas GUS sebagian besar tidak ada di rizoid, kecuali sinyal lemah yang diamati dalam satu garis transgenik (garis #2) (Gbr. 4a ). Pada irisan melintang dan membujur talus dewasa, aktivitas GUS dideteksi di sepanjang pelepah daun pada sisi ventral talus (Gbr. 4b,c ). Selain itu, pengamatan histologis cawan gemma menunjukkan pewarnaan GUS baik di gemmae maupun pangkal cawan gemma (Gbr. 4d ). Hasil ini menunjukkan bahwa Mp RLF diekspresikan dalam talus dan selama pembentukan gemma. Lebih jauh lagi, dalam anteridiofor, aktivitas GUS diamati dalam cakram anteridia dan bagian-bagian tangkai (Gbr. 4e ). Pengamatan ini konsisten dengan data ekspresi yang tersedia untuk umum dari Gambar S3 , yang selanjutnya mendukung bahwa Mp RLF diekspresikan dalam berbagai organ selama fase pertumbuhan vegetatif dan reproduksi.

Protein MpRLF terlokalisasi di sitosol
Pada A. thaliana , RLF terlokalisasi pada sitosol (Ikeyama et al ., 2010 ). Untuk menyelidiki lokalisasi subseluler MpRLF, garis Mp E2Fpro:XVE> Mp RLFcds-GFP yang dijelaskan di atas digunakan untuk pengamatan. Pada sel epidermis dekat takik apikal gemmaling Mp E2Fpro::XVE> Mp RLFcds-GFP yang tumbuh selama 24 jam pada medium yang mengandung EST, MpRLF-GFP terlokalisasi pada sitosol dan tidak terlokalisasi bersama kloroplas, nukleus, atau membran plasma (Gbr. 4f ), yang konsisten dengan lokalisasi subseluler protein Arabidopsis RLF (RLF-GFP) (Ikeyama et al ., 2010 ). Hasil ini menunjukkan bahwa MpRLF berfungsi dalam sitosol.
Mp RLF sangat penting untuk pembentukan talus dan cangkir gemma yang tepat
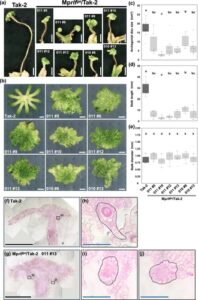
Untuk menyelidiki peran biologis Mp RLF , kami menghasilkan mutan Mp RLF (Mp rlf ge ) menggunakan metode CRISPR-Cas9. Urutan target CRISPR-Cas9 dirancang di persimpangan intron kedua dan ekson ketiga, hulu Cytb5-HBD (Gbr. 5a ). Analisis sekuensing mutan yang diperoleh mengungkapkan delesi -2, -18, dan -1 bp di wilayah genomik dekat situs target pada galur independen (Mp rlf ge #3, #9, dan #11), masing-masing (Gbr. 5b ). Selain itu, delesi ini menyebabkan pergeseran bingkai dalam urutan mRNA ini, yang menyebabkan kodon stop prematur terjadi di hulu Cytb5-HBD (Gbr. 5c ). Ketiga alel mutan Mp rlf ge menunjukkan retardasi pertumbuhan dibandingkan dengan WT (Gbr. 5d,e , S4a ). Tanaman WT membentuk gemma cup dengan gemmae pada permukaan dorsal talus, sementara pada ketiga mutan Mp rlf ge , pembentukan gemma cup yang tepat dihambat dan struktur seperti gemma terbentuk langsung pada permukaan talus (Gbr. 6a ). Uji komplementasi dengan urutan pengkodean Mp RLF dengan mutasi sinonim resisten CRISPR (Mp RLFcds_resistant ; Gbr. S4b ) menunjukkan bahwa tanaman transgenik yang mengekspresikan Mp RLFcds_resistant di bawah kendali promotor Mp RLF pada latar belakang Mp rlf ge tumbuh sebaik WT dan membentuk gemma cup yang tepat (Gbr. 5d,e ). Hasil ini menunjukkan bahwa fenotipe yang diamati pada Mp rlf ge (keterbelakangan pertumbuhan talus dan gangguan pembentukan gemma cup) disebabkan oleh mutasi loss-of-function pada gen Mp RLF , dengan demikian mengonfirmasi bahwa Mp RLF penting untuk pertumbuhan talus yang tepat dan pembentukan gemma cup.

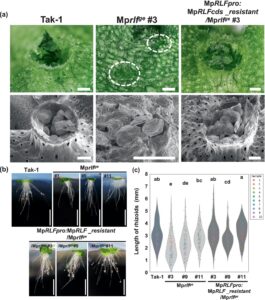
Lebih jauh, perbandingan pemanjangan rizoid di antara WT, mutan, dan galur pelengkap mengungkapkan bahwa mutan Mp rlf ge menunjukkan panjang rizoid keseluruhan yang sedikit lebih pendek (Gbr. 6b,c ). Akan tetapi, beberapa individu diamati memiliki rizoid yang panjangnya sebanding dengan WT, dan tidak terdeteksi kelainan signifikan dalam perkembangan rizoid. Hasil ini, bersama dengan ekspresi GUS lemah yang diamati dalam rizoid, menunjukkan bahwa Mp RLF mungkin memainkan peran kecil dalam pembentukan rizoid.
Berikutnya, kami menginduksi pembentukan organ reproduksi di bawah cahaya merah jauh dan cahaya putih dan mengamati perkembangan anteridiofor. Pada galur WT dan galur komplementasi, anteridiofor yang mengandung anteridia terbentuk, sedangkan pada mutan Mp rlf ge , anteridiofor tampak lebih pendek, dan bentuk cakram anteridia berubah (Gbr. 7a,b ). Analisis ukuran cakram anteridia, panjang tangkai, dan diameter tangkai menunjukkan bahwa ukuran cakram pada mutan cocok dengan WT, tetapi tangkainya lebih pendek dan lebih tebal (Gbr. 7c–e ). Selain itu, kami mengamati bahwa pada Mp rlf ge , struktur seperti anteridium terekspos pada permukaan cakram anteridia, sedangkan pada tanaman WT, struktur ini biasanya tertutup di dalam cakram (Gbr. 7f,g ). Selain itu, kami menghasilkan beberapa galur mutan Mp rlf ge pada latar belakang Tak-2 betina menggunakan metode CRISPR-Cas9 (Gbr. S4c ). Ketika organ reproduksi diinduksi dalam mutan ini, arkegoniofor lebih pendek tingginya daripada WT dan membentuk cakram arkegonial tidak teratur yang tidak memiliki tonjolan normal (Gbr. 8a,b ). Pengukuran luas cakram arkegonial, panjang tangkai, dan diameter tangkai mengungkapkan bahwa mutan memiliki luas cakram arkegonial yang lebih kecil dan tangkai yang lebih panjang daripada WT, sementara tidak ada perbedaan signifikan yang diamati dalam diameter tangkai (Gbr. 8c–e ). Kelainan pada organ reproduksi tanaman jantan dan betina ini diamati bahkan pada tahap awal perkembangan organ reproduksi (Gbr. S4d ). Lebih jauh, penampang melintang arkegoniofor mengungkapkan bahwa mutan membentuk struktur seperti arkegonium yang abnormal dan tertutup sepenuhnya, bukan arkegonium khas yang terlihat pada WT (Gbr. 8f–j ). Hasil ini menunjukkan bahwa MpRLF mengatur banyak aspek perkembangan organ reproduksi, termasuk pertumbuhan tangkai dan proporsi struktural.

Untuk menyelidiki dampak dari ekspresi berlebih Mp RLF pada perkembangan organ, kami menghasilkan tanaman transgenik dengan ekspresi Mp RLFcds-GFP yang dapat diinduksi oleh EST di bawah kendali Mp E2Fpro (Mp E2Fpro:XVE>> Mp RLFcds-GFP ). Menariknya, sementara mutan kehilangan fungsi menunjukkan area talus yang berkurang, ekspresi berlebih MpRLF yang diinduksi EST (MpRLF-GFP) secara jelas menekan pertumbuhan talus (Gbr. S5 ). Hasil-hasil ini menunjukkan bahwa regulasi ekspresi Mp RLF yang tepat penting untuk pertumbuhan vegetatif M. polymorpha . Secara keseluruhan, hasil-hasil ini menunjukkan bahwa Mp RLF diperlukan untuk pembentukan organ yang tepat selama pertumbuhan vegetatif dan reproduktif M. polymorpha .
Arabidopsis thaliana RLF sebagian melengkapi fenotipe mutan Mp rlf ge di M. polymorpha
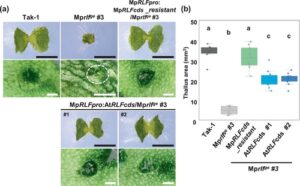
Untuk memeriksa konservasi fungsional RLF antara A. thaliana dan M. polymorpha , kami melakukan uji komplementasi menggunakan mutan Mp rlf ge #3 dan menemukan bahwa galur yang mengekspresikan A. thaliana RLF di bawah promotor MpRLF (Mp RLFpro: Pada RLFcds/ Mp rlf ge #3) menunjukkan penyelamatan parsial fenotipe mutan dalam morfologi talus dan pembentukan cawan gemma (Gbr. 9 ). Hasil-hasil ini dan fakta bahwa Mp RLF menyelamatkan fenotipe pembentukan LR dari mutan A. thaliana rlf (Gbr. 3c,d ) mengindikasikan bahwa Mp RLF dan A. thaliana RLF berbagi fungsi molekuler yang dilestarikan dan umum yang penting untuk perkembangan organ.
MpRLF sangat mengubah ekspresi gen yang berhubungan dengan metabolisme
Akhirnya, untuk mendapatkan wawasan tentang jalur yang dilalui MpRLF dalam mengatur pertumbuhan dan perkembangan organ, kami melakukan analisis RNA sequencing (RNA-Seq) menggunakan Tak-1 dan mutan Mp rlf ge (Mp rlf ge #3). Dari sekuens yang diperoleh dari pembacaan bersih, 97,96% dapat dipetakan ke genom M. polymorpha (Marpolbase MpTak_v6.1r2; Bowman et al ., 2017 ; Montgomery et al ., 2020 ). Ada 3734 gen yang diekspresikan secara diferensial (DEG) dalam Mp rlf ge , dengan 1839 mengalami peningkatan regulasi dan 1895 mengalami penurunan regulasi (Gbr. 10a ).

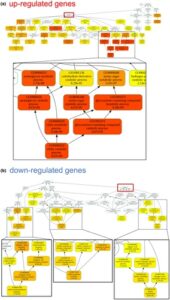
Untuk mengkarakterisasi variasi ekspresi gen dalam Mp rlf ge dengan lebih baik , kami melakukan analisis pengayaan GO. Hasilnya menunjukkan bahwa proses biologis yang paling terpengaruh dalam Mp rlf ge adalah proses seluler (GO:0009987) dan proses metabolisme (GO:0008152). Dalam komponen seluler, proses yang paling terpengaruh adalah bagian sel (GO:0044464), sel (GO:0005623), membran (GO:0016020), bagian membran (GO 0044425), organel (GO:0043226), dan bagian organel (GO:0044446). Dalam fungsi molekuler, pengikatan (GO:0005488) dan aktivitas katalitik (GO:0003824) adalah proses yang paling banyak diinduksi (Gbr. S6a ). Lebih jauh, analisis jalur KEGG dari DEG menunjukkan bahwa mereka diperkaya untuk istilah terkait metabolisme (Gbr. S6b ). Selain itu, analisis pengayaan set gen (GSEA) menggunakan diagram gelembung menunjukkan bahwa, di antara proses biologis, proses yang terkait dengan proses metabolisme – seperti proses metabolisme asam nukleat (GO:0090304), proses metabolisme sekunder (GO:0019748), dan proses metabolisme asam karboksilat (GO:0019752) – diperkaya dalam gen yang diatur naik (Gbr. 10b ). Dalam analisis yang sama untuk gen yang diatur turun, proses metabolisme senyawa nitrogen seluler (GO:0034641) khususnya diperkaya. Temuan ini menunjukkan bahwa keadaan metabolisme berubah secara drastis dalam Mp rlf ge (Gbr. 10c ). Analisis pengayaan set gen, berdasarkan fungsi molekuler, menunjukkan bahwa istilah GO yang terkait dengan oksidoreduktase, seperti aktivitas oksidoreduktase (GO:0016491) dan aktivitas katekol oksidase (GO:0004097), diperkaya dalam gen yang diatur naik, dan aktivitas transporter transmembran (GO:0022857) dan pengikatan heme (GO:0020037) diperkaya dalam gen yang diatur turun (Gbr. S7a ). Analisis pengayaan set gen, berdasarkan komponen seluler, mengungkapkan bahwa gen yang diatur naik diperkaya dalam apoplas (GO:0048046) dan kompleks yang mengandung protein nuklir (GO:0140513), sementara gen yang diatur turun diperkaya dalam kompleks yang mengandung protein (GO:0032991) dan membran plasma (GO:0005886) (Gbr. S7b ). Selain itu, kami melakukan analisis hierarki GO dari proses biologis pada 200 gen teratas yang diatur naik dan diatur turun. Gen yang mengalami peningkatan regulasi diperkaya untuk istilah GO yang terkait dengan metabolisme kitin (Gbr. 11a ; Tabel S2 ). Untuk gen yang mengalami penurunan regulasi, istilah GO yang terkait dengan metabolisme nitrat, transportasi, dan respons diperkaya (Gbr. 11b ; Tabel S2 ). Hasil ini menunjukkan bahwa pertumbuhan dan perkembangan organ abnormal yang diamati pada Mp rlf ge mungkin disebabkan oleh perubahan dalam status metabolik, yang berpotensi melibatkan MpRLF sebagai protein Cytb5-HBD.
Diskusi
Dalam studi ini, kami menunjukkan bahwa A. thaliana RLF adalah HBP dan mengonfirmasi bahwa dua residu histidin pada posisi 161 dan 184 dalam Cytb5-HBD diperlukan untuk pengikatan heme secara in vitro dan untuk fungsi RLF pada tanaman . Ortolog RLF sangat terkonservasi pada berbagai tanaman hijau, dan dua residu histidin, yang penting untuk pengikatan heme dalam Cytb5-HBD, ditemukan pada ortolog RLF. Hasil ini menyiratkan bahwa ortolog RLF pada tanaman lain juga mengikat heme melalui dua residu histidin dan bahwa kemampuan pengikatan heme dari Cytb5-HBD diperlukan untuk fungsi biologis protein RLF. Lebih jauh, kami menunjukkan bahwa (1) MpRLF, sebuah ortolog RLF di M. polymorpha , juga mengikat heme in vitro , bahwa (2) Mp RLF menyelamatkan fenotipe mutan pembentukan LR dari mutan rlf A. thaliana , dan bahwa (3) Mp RLF sangat penting untuk perkembangan organ yang tepat di M. polymorpha . Hasil-hasil ini menunjukkan bahwa ortolog RLF di tanaman lain mungkin juga berperan dalam perkembangan organ. Oleh karena itu, akan menarik untuk memverifikasi apakah ortolog RLF dari berbagai tanaman dan alga, termasuk charophytes, bryophytes, lycophytes, gymnospermae, angiospermae, serta alga hijau dan merah, mampu melengkapi mutan rlf di A. thaliana atau mutan Mp rlf ge di M. polymorpha , atau keduanya. Ini akan memberikan wawasan ke dalam konservasi evolusi fungsi RLF di seluruh tanaman dan alga.
Analisis transkriptom menggunakan mutan Mp rlf ge juga menunjukkan bahwa MpRLF dapat berkontribusi pada perkembangan organ dengan mengatur beberapa jalur metabolisme. Analisis pengayaan Gene Ontology dan jalur KEGG mengungkapkan bahwa variasi dalam ekspresi gen dalam Mp rlf ge paling menonjol dalam proses metabolisme, komponen seluler, dan fungsi molekuler. Lebih jauh lagi, hierarki GO dari 200 gen yang mengalami penurunan regulasi teratas diperkaya dalam istilah GO yang terkait dengan respons nitrat, transportasi, dan metabolisme (Gbr. 11b ; Tabel S2 ). Yang menarik adalah fakta bahwa RLF mengatur pembentukan LR di A. thaliana (Ikeyama et al ., 2010 ). Hubungan antara pembentukan LR dan nitrat telah dipelajari secara ekstensif dalam beberapa tahun terakhir. Modul pengatur OBP4-XTH9 yang responsif terhadap nitrat memainkan peran penting dalam perkembangan LR dan mengatur pertumbuhan LR sebagai respons terhadap perubahan konsentrasi nitrat (Xu & Cai, 2019 ). Lebih jauh, telah ditunjukkan bahwa nitrat berinteraksi dengan auksin selama pembentukan LR, dan bahwa distribusi konsentrasi nitrat secara signifikan memengaruhi perkembangan LR (Pélissier et al ., 2021 ). Kami belum meneliti keterlibatan nitrat dalam perkembangan organ yang bergantung pada RLF, tetapi mungkin ada hubungan kausal antara nitrat dan perkembangan organ abnormal pada Mp rl fge .
Mengenai keluarga A. thaliana Cytb5-HBD, protein Cytb5-HBD berfungsi terutama sebagai pembawa elektron, mentransfer elektron dari reduktase ke protein target. Secara khusus, CB5D menyediakan elektron ke enzim yang mengubah prekursor sintesis G-lignin menjadi prekursor sintesis S-lignin, dengan demikian mengatur sintesis lignin (Gou et al ., 2019 ). CB5B terlibat dalam sintesis sphingolipid melalui interaksinya dengan FAH1, yang menghidroksilasi posisi C-2 asam lemak dan menyediakan elektron yang diperlukan untuk reaksi (Nagano et al ., 2012 ). Temuan ini menunjukkan bahwa RLF juga dapat berfungsi sebagai pemancar elektron. Konsisten dengan ini, analisis RNA-seq kami menunjukkan bahwa ekspresi gen yang terlibat dalam transfer elektron dan redoks diubah dalam Mp rlf g , menunjukkan bahwa MpRLF adalah transferase elektron yang mengatur keadaan redoks seluler. Selain itu, protein Cytb5-HBD juga dapat menjadi transferase elektron dalam M. polymorpha , meskipun jalur aksi spesifiknya masih harus dijelaskan. Oleh karena itu, mengidentifikasi protein yang berinteraksi dengan MpRLF melalui ko-imunopresipitasi atau pelabelan kedekatan menggunakan ligase biotin aktif (TurboID) akan sangat berguna (Mair et al ., 2019 ; Zhang et al ., 2020 ).
Mutasi Mp rlf ge menunjukkan penghambatan pembentukan gemma cup dan pertumbuhan talus, yang konsisten dengan aktivitas promoter Mp RLF di dasar gemma cup dan dekat meristem talus (Gbr. 4-6 ) . Hasil ini mengindikasikan bahwa Mp RLF penting untuk pembentukan talus dan gemma cup yang tepat. Beberapa gen telah diidentifikasi sebagai penting untuk pembentukan gemma cup dan gemma. Gen GEMMA CUP-ASSOCIATED MYB1 ( GCAM1 ), yang mengkode faktor transkripsi tipe R2R3-MYB, merupakan pengatur utama pembentukan gemma cup, dan galur knockout GCAM1 tidak dapat membentuk gemma cup dan gemma (Yasui et al ., 2019 ). Gen KARAPPO , yang mengkode faktor pertukaran nukleotida guanin ROP (RopGEF), juga penting untuk pembentukan gemma. Mutan loss-of-function dari KARAPPO menampilkan fenotipe khas dari gangguan pembentukan gemma tanpa mempengaruhi pembentukan gemma cup (Hiwatashi et al ., 2019 ). Selain itu, mutan gen Mp RKD , yang mengatur diferensiasi dan inisiasi sel germinal, cacat dalam membentuk tepi gemma cup meskipun gemma masih berkembang dari dasar gemma cup (Koi et al ., 2016 ; Rövekamp et al ., 2016 ). Dibandingkan dengan mutan di atas, Mp rlf ge membentuk gemma pada talus sementara pembentukan gemma cup yang tepat dihambat. Menariknya, dalam dataset RNA-seq, ekspresi Mp5g06550/KARAPPO tidak berubah dalam Mp rlf ge , sementara ekspresi Mp3g04030/ Mp RKD dan Mp6g04830/GCAM1 menurun (Tabel S3 ). Perlu dicatat bahwa kami menggunakan tanaman yang tumbuh selama 16 hari, yang merupakan waktu yang cukup untuk pembentukan cawan gemma pada tanaman WT dalam kondisi pertumbuhan kami. Dengan mempertimbangkan hal-hal ini, proses metabolisme yang terkait dengan MpRLF dapat berkontribusi pada ekspresi GCAM1 dan Mp RKD yang tepat selama proses pembentukan cawan gemma. Selain cacat dalam perkembangan vegetatif, mutan Mp rlf ge menunjukkan perkembangan abnormal pada organ reproduksi jantan dan betina, yang konsisten dengan aktivitas promotor Mp RLF pada organ reproduksi (Gbr. 4 , 7 , 8 ). Hasil-hasil ini menunjukkan bahwa Mp RLFjuga penting untuk perkembangan organ reproduksi yang tepat. Analisis lebih lanjut tentang distribusi spasial dan pola ekspresi spesifik tahap gen yang mengatur pembentukan cawan gemma, gemma, dan organ reproduksi (anteridiofor dan arkegoniofor) dalam Mp rlf ge akan menjelaskan bagaimana Mp RLF mengatur perkembangan organ-organ ini.
Area talus yang berkurang yang diamati pada garis ekspresi berlebih, meskipun mewakili kondisi genetik yang berlawanan dengan mutan kehilangan fungsi, merupakan hasil yang tidak terduga (Gbr. S5 ). Kami mengusulkan bahwa ekspresi berlebih Mp RLF dapat mengubah keadaan metabolisme intraseluler, mengganggu keseimbangan proses seluler seperti pembelahan dan perluasan sel, yang pada akhirnya menyebabkan cacat pertumbuhan. Analisis Gene Ontology dari data RNA-seq dari mutan Mp RLF mengungkapkan pengayaan dalam apoplas (GO:0048046) dan membran plasma (GO:0005886), yang menunjukkan perubahan pada sifat membran. Ini menyiratkan bahwa perubahan serupa dalam dinamika membran dapat terjadi pada garis ekspresi berlebih, yang berpotensi memengaruhi jalur transduksi sinyal yang mengatur perkembangan dan perluasan siklus sel. Memeriksa ekspresi gen yang terlibat dalam regulasi siklus sel (misalnya CYC , CDK , dan E2F ) dan perluasan sel (misalnya EXP , CESA , dan XTH ) pada garis ekspresi berlebih Mp RLF dapat memberikan wawasan tentang mekanisme yang mendasari cacat pertumbuhan ini. Ekspresi berlebihan protein heme/steroid-binding domain (Cytb5-HBD) mirip sitokrom b 5 telah dilaporkan menginduksi perubahan metabolik. Misalnya, dalam Malus domestica (apel), ekspresi berlebihan Md CYB5 disertai dengan peningkatan regulasi gen biosintesis antosianin, seperti Md ANR , Md CHI , Md DFR , dan Md UF3GT , yang mengarah pada peningkatan signifikan dalam akumulasi antosianin (Zhang et al ., 2024 ). Demikian pula, dalam ekspresi berlebihan protein Cytb5-HBD MpRLF, sangat mungkin bahwa gen yang terlibat dalam metabolisme intraseluler juga diubah. Untuk mendapatkan wawasan yang lebih dalam tentang mekanisme ini, penelitian masa depan harus fokus pada mengidentifikasi jaringan gen yang diatur oleh MpRLF melalui pendekatan seperti RNA-seq dan profil metabolom menggunakan garis ekspresi berlebihan.
Analisis filogenetik mengungkapkan bahwa ortolog RLF membentuk klade yang berbeda dan didukung dengan baik, terpisah dari keluarga protein Cytb5-HBD lainnya seperti CB5B/D, CB5E, dan CB5L (Gbr. 2 ). Ini menunjukkan bahwa RLF mewakili subkelompok yang terspesialisasi secara fungsional dalam keluarga Cytb5-HBD. Konservasi ortolog RLF di seluruh tanaman yang beragam, termasuk angiospermae, gymnospermae, pakis, lumut hati, dan lumut, menunjukkan bahwa fungsi RLF telah dilestarikan sepanjang evolusi tanaman darat. Lebih jauh lagi, keberadaan ortolog RLF dalam alga streptofit, seperti Chara (charophytes), dan alga hijau, termasuk Chlamydomonas dan Volvox , menunjukkan bahwa asal-usulnya mendahului munculnya tanaman darat. Distribusi yang lebih luas ini menimbulkan kemungkinan bahwa RLF awalnya memainkan peran mendasar dalam proses seluler atau perkembangan yang kemudian diadopsi untuk pengembangan organ tanaman darat. Mengingat bahwa penelitian kami berfokus pada peran MpRLF dalam perkembangan organ, akan menarik untuk menyelidiki apakah fungsi leluhur RLF pada alga terkait dengan pembelahan dan diferensiasi sel, respons stres, atau mekanisme seluler fundamental lainnya. Sebagian besar spesies tumbuhan memiliki satu ortolog RLF, yang menyiratkan kemungkinan diversifikasi fungsional atau subfungsionalisasi. Selain itu, RLF A. thaliana mampu melengkapi mutan Mp rlf ge , yang menunjukkan bahwa regulasi perkembangan organ yang dimediasi RLF telah dilestarikan di seluruh tumbuhan darat. Temuan-temuan ini menunjukkan bahwa perkembangan organ yang dimediasi RLF yang bergantung pada pengikatan heme tetap menjadi fitur yang stabil dan penting sepanjang evolusi tumbuhan darat. Di sisi lain, daerah tak teratur N-terminal RLF bervariasi di antara spesies tumbuhan yang berbeda (Gbr. S2 ). Meskipun kami tidak menganalisis daerah tak teratur secara rinci dalam penelitian ini, kami tidak dapat mengesampingkan bahwa struktur N-terminal juga memengaruhi fungsi RLF. Telah ditunjukkan bahwa sebagian dari daerah N-terminal RLF padi mirip dengan struktur N-terminal NADH sitokrom b 5 oksidoreduktase manusia (Ncb5or) dan mengadopsi struktur heliks ketika berinteraksi dengan Cytb5-HBD dalam protein yang sama (Benson et al ., 2024 ). Bagaimana struktur heliks berkontribusi pada fungsi RLF saat ini tidak diketahui, tetapi perubahan yang bergantung pada Cytb5-HBD pada daerah yang tidak teratur mungkin signifikan. Sementara struktur N-terminal setiap spesies tanaman berbeda, MpRLF menyelamatkan fenotipe pembentukan LR dari mutan rlf A. thaliana , yang menunjukkan bahwa daerah yang tidak teratur mungkin tidak penting untuk fungsi RLF. Hasil-hasil ini menimbulkan pertanyaan lebih lanjut tentang signifikansi daerah yang tidak teratur dalam keluarga protein RLF.
Studi kami menyoroti peran penting aktivitas pengikatan heme dalam fungsi RLF dan kebutuhannya untuk perkembangan organ yang tepat di A. thaliana dan M. polymorpha . Kami berhipotesis bahwa RLF yang terikat heme bertindak sebagai mediator pengatur, yang memungkinkan RLF untuk berpartisipasi dalam reaksi redoks atau interaksi protein yang penting untuk transisi perkembangan. Konservasi evolusi fungsi RLF di seluruh tanaman darat menggarisbawahi pentingnya pengikatan heme dalam perkembangan organ yang kompleks. Studi lebih lanjut tentang ortolog RLF, termasuk A. thaliana RLF dan Mp RLF , akan memperdalam pemahaman kita tentang hubungan antara evolusi Cytb5-HBD dan peran biologis dari keluarga protein RLF, yang memajukan pengetahuan kita tentang mekanisme yang dilestarikan dalam perkembangan tanaman.




Tinggalkan Balasan