Abstrak
Strain penentuan jenis kelamin genetik (GSS) adalah alat penting untuk teknik serangga steril (SIT), metode pengendalian hama serangga yang ramah lingkungan dan khusus spesies. GSS menampilkan fenotipe khusus jenis kelamin, yang memungkinkan penyortiran jenis kelamin di fasilitas pemeliharaan massal dan pelepasan khusus jantan, yang secara signifikan meningkatkan efektivitas biaya dan efisiensi program SIT. Dalam GSS klasik, hubungan jenis kelamin gen penanda, seperti pupa putih ( wp ), dicapai melalui translokasi yang diinduksi iradiasi antara autosom pembawa penanda dan kromosom Y. Namun, pendekatan ini dapat membuat jantan GSS semisteril. Konsep GSS neo-klasik yang baru-baru ini diusulkan menyarankan penggunaan penyuntingan genom untuk mencapai hubungan jenis kelamin dengan memasukkan langsung alel penanda tipe liar ke kromosom Y, yang berpotensi menghasilkan jantan GSS dengan kesuburan yang lebih tinggi. Dalam penelitian ini, kami memeriksa gen Ceratitis capitata wp sebagai penanda genetik untuk konsep GSS neo-klasik dan mengembangkan versi minimal tanpa intron dari gen ini, yang disebut mini- wp . Kami menunjukkan bahwa satu salinan mini- wp cukup untuk memulihkan fenotipe puparium coklat tipe liar dan berfungsi saat terintegrasi pada berbagai posisi dalam genom C. capitata , termasuk kromosom X. Karena ukurannya yang lebih kecil (4689 bp, termasuk 2000 bp wilayah promotor putatif) relatif terhadap alel wp tipe liar penuh (20868 bp), mini- wp dapat memfasilitasi penyisipan yang tepat ke dalam kromosom Y, yang merupakan langkah penting menuju terwujudnya GSS neo-klasik. Lebih jauh, metodologi yang dikembangkan untuk merancang dan menguji mini- wp pada lalat buah dapat diadaptasi ke spesies Tephritid lain dengan gen wp yang teridentifikasi .
Perkenalan
Strain penentuan jenis kelamin genetik (GSS) adalah alat yang terbukti berharga untuk penyortiran jenis kelamin di fasilitas pemeliharaan massal serangga dan elemen kunci untuk penerapan teknik serangga mandul (SIT) yang hemat biaya untuk pengendalian hama (Franz et al. , 2021 ; Klassen et al. , 2021 ; Mumford, 2021 ). Program SIT didasarkan pada pemeliharaan massal serangga target, diikuti dengan sterilisasi dan pelepasannya ke area yang terinfestasi. Pejantan mandul kawin dengan betina liar, sehingga tidak menghasilkan keturunan, sehingga secara bertahap mengurangi populasi serangga di area yang diobati (Knipling, 1955 ). GSS secara klasik memerlukan dua komponen: penanda mutasi yang dapat dipilih yang memungkinkan pemisahan jenis kelamin atau eliminasi betina pada tahap sedini mungkin, dan hubungan alel tipe liar (WT) dari penanda ini dengan jenis kelamin jantan. Contoh GSS yang terkenal dan berhasil adalah VIENNA-8, yang dikembangkan pada Ceratitis capitata (Wiedemann) (Diptera: Tephritidae), yang umumnya dikenal sebagai lalat buah Mediterania, atau medfly (Franz et al. , 1994 ; Augustinos et al. , 2017 ; Franz et al. , 2021 ). GSS ini membawa dua penanda yang dapat dipilih, pupa putih ( wp − ) dan letal peka suhu ( tsl − ), yang awalnya terletak pada autosom (kromosom 5). Translokasi yang diinduksi iradiasi telah memindahkan alel WT dari kedua penanda tersebut ke kromosom Y, yang memfasilitasi pemisahan jenis kelamin; lalat betina, homozigot untuk alel yang bermutasi, muncul dari puparia putih dan peka terhadap suhu tinggi. Sebaliknya, lalat jantan heterozigot muncul dari puparia coklat dan tidak terpengaruh oleh suhu tinggi ini. Biasanya, selama operasi di fasilitas pemeliharaan massal, embrio betina tidak bertahan hidup dalam perawatan pada suhu 34 °C selama 24 jam, sedangkan embrio jantan tidak terpengaruh (Franz et al. , 2021 ).
Akan tetapi, pembentukan galur GSS merupakan proses yang membutuhkan banyak sumber daya, yang utamanya diatur oleh faktor stokastik. Misalnya, fenotipe wp − pada Tephritidae, yang muncul dari mutasi alami, sejauh ini hanya diisolasi pada lalat buah (Rössler, 1979 ), lalat melon ( Zeugodacus cucurbitae ) (McInnis et al. , 2004 ), dan lalat buah oriental ( Bactrocera dorsalis ) (McCombs & Saul, 1992 , 1995 ). Lebih jauh lagi, GSS berdasarkan translokasi alel WT autosomal ke kromosom Y mungkin, tergantung pada titik henti translokasi kromosom Y dan perilaku segregasi yang dihasilkan selama meiosis jantan, bersifat semisteril dan tidak stabil secara genetik, yaitu, rekombinasi autosomal mungkin terjadi selama pemeliharaan massal, yang berpotensi membahayakan GSS (Franz et al. , 2021 ; Cáceres et al. , 2023 ). Oleh karena itu, pengembangan alat umum untuk pengembangan GSS yang lebih cepat tetap diperlukan untuk meningkatkan dan memperluas SIT ke spesies serangga lainnya (Bourtzis & Vreysen, 2021 ).
Meskipun fenotipe wp − telah digunakan dalam GSS selama beberapa dekade, dasar genetik untuk fenotipe ini di Tephritids baru-baru ini terungkap (Ward et al. , 2021 ), yang memungkinkan terciptanya beberapa garis berbasis CRISPR/Cas dengan fenotipe wp − di lalat buah mediterania, lalat buah Queensland, B. tryoni , dan lalat melon (Ward et al. , 2021 ; Paulo et al. , 2022 ). Identifikasi gen penanda yang sesuai ini dan tambahan (Robinson, 2002 ; Chen et al. , 2022 ; Sollazzo et al. , 2024 ; Paulo et al. , 2025 ), dikombinasikan dengan upaya berkelanjutan untuk menghasilkan genom referensi lengkap berkualitas tinggi dari artropoda yang menarik bagi pertanian dan kesehatan masyarakat (Matthews et al. , 2018 ; Palatini et al. , 2020 ; Childers et al. , 2021 ; Fisher et al. , 2022 ; Wang et al. , 2023 ; Zhang et al. , 2023 ), dan kemajuan terbaru dalam penyuntingan genetik pada serangga menggunakan teknik molekuler modern (Aumann et al. , 2018 ; Buchman & Akbari, 2019 ; Meccariello et al. , 2019 ; Aumann et al. , 2020 ; Gamez et al. , 2021 ; Häcker et al. , 2021 ; Yan et al. , 2023 ), memungkinkan konsep “pendekatan genetik neo-klasik” untuk menghasilkan GSS. Dalam pendekatan ini, hubungan jenis kelamin dapat dicapai dengan memasukkan alel WT dari penanda yang dapat dipilih ke dalam kromosom Y atau di dekat faktor penentu jantan dalam galur dengan penanda yang bermutasi (Nguyen et al. , 2021 ; Yan et al. , 2023 ; Yan et al. , 2024 ). Karena efisiensi penyisipan yang dimediasi oleh perbaikan yang diarahkan oleh homologi CRISPR/Cas (HDR) sensitif terhadap ukuran penyisipan dan dapat ditingkatkan dengan kargo yang lebih kecil (Li et al. , 2014 ; Paix et al. , 2017), menggunakan rekayasa konstruksi gen minimal alih-alih alel endogen penuh dapat meningkatkan keberhasilan penyuntingan. Hal ini mungkin relevan khususnya untuk pengembangan GSS neo-klasik, di mana penargetan kromosom Y yang miskin gen dan sangat repetitif diperlukan (Charlesworth & Charlesworth, 2000 ; Bachtrog, 2013 ; Choo et al. , 2019 ).
Di sini, kami meneliti gen wp sebagai penanda genetik untuk konsep GSS neo-klasik di C. capitata , dan melaporkan keberhasilan pemulihan fenotipe pupa coklat WT di lalat buah melalui integrasi piggyBac versi tanpa intron dari gen pupa putih ke dalam galur wp − .
Bahan dan metode
Semua primer yang digunakan dalam penelitian ini dirancang dengan Geneious Prime (versi 2021.2.2; Kearse et al. , 2012 ) dan tercantum dalam Tabel S1 .
Strain serangga dan pemeliharaannya
Strain Ceratitis capitata (Wiedemann) tipe liar Egypt-II (EgII), mata putih ( we − ), dan pupa putih ( wp − ) diperoleh dari Laboratorium Pengendalian Hama Serangga, Pusat Teknik Nuklir Gabungan FAO/IAEA dalam Pangan dan Pertanian (IPCL/IAEA, Seibersdorf, Austria). Strain wp − yang dimodifikasi CRISPR ( wp −(CRISPR) ) sebelumnya telah dibuat di laboratorium (strain Cc_D ) (Ward et al. , 2021 ). Strain yang homozigot ganda untuk mutasi alami mata putih dan pupa putih ( we − wp − / we − wp − ) diproduksi dengan cara mengawinkan galur mata putih yang bermutasi alami dan galur pupa putih yang bermutasi alami . Semua galur serangga dibesarkan dalam kondisi laboratorium standar pada suhu 25 ± 1 °C, kelembaban relatif (RH) 48%, dan siklus terang/gelap 14 jam : 10 jam. Larva dibesarkan dengan makanan larva berbasis wortel yang dibuat dengan 1,4 kg wortel beku yang dimasak, 16 g natrium benzoat (VWR International GmbH, Darmstadt, Jerman), 500 g bubuk wortel (Van Drunen Farms, Momence, IL, AS), 168 g enzimatik hidrolisat ragi (MP Biomedicals, Solon, OH, AS), 20 mL larutan asam klorida 25% (v/v) (Carl Roth GmbH + Co. KG, Karlsruhe, Jerman), dicampur dengan sekitar 2 L air suling untuk menyesuaikan kekentalan. Tikus dewasa diberi makan sepuasnya dengan campuran gula dan ragi hidrolisat enzimatik (MP Biomedicals) (3 : 1, v : v), dan air.
Amplifikasi cepat ujung cDNA
Total RNA diekstraksi dari satu prepupa tunggal galur EgII menggunakan kit Monarch total RNA Miniprep (New England Biolabs Inc., Ipswich, MA, AS) mengikuti petunjuk pabrik pembuatnya. 5 µ g total RNA bebas DNA digunakan untuk mengisolasi mRNA menggunakan Modul Isolasi Magnetik mRNA NEBNext Poly(A) (NEB #E7490, New England Biolabs Inc., Ipswich, MA, AS). Amplifikasi cepat ujung cDNA 5′ dan 3′ (RACE) dilakukan menggunakan Kit SMARTer ® RACE 5’/3′ (Takara Bio USA, Inc., Mountain View, CA, AS) mengikuti petunjuk pabrik pembuatnya dengan primer P2125 dan P2126. Kondisi siklusnya adalah sebagai berikut: 5 siklus 94 °C selama 30 detik, 72 °C selama 3 menit, 5 siklus 94 °C selama 30 detik, 70 °C selama 30 detik, 72 °C selama 3 menit, 25 siklus 94 °C selama 30 detik, 65 °C selama 30 detik, 72 °C selama 3 menit. Produk PCR dianalisis melalui elektroforesis gel, diekstraksi menggunakan Zymoclean Gel DNA Recovery Kit (Zymo Research Europe GmbH, Freiburg, Jerman), dikloning ke dalam vektor pRACE yang dilinearisasi (Takara Bio USA, Inc.), dan ditransformasikan ke dalam sel XL1-Blue MR Supercompetent ( E. coli Δ(mcrA)183 Δ(mcrCB-hsdSMR-mrr)173 endA1 supE44 thi-1 recA1 gyrA96 relA1 lac [F proAB lacIqZΔM15 Tn10 (Tetr)]; Agilent Technologies, Santa Clara, CA, AS). Koloni individu ditumbuhkan dalam medium LB yang mengandung 100 ng/ µ L ampisilin (Carl Roth GmbH + Co. KG, Karlsruhe, Jerman) dan plasmid diekstraksi menggunakan kit NucleoSpin Plasmid Mini (Macherey-Nagel GmbH & Co. KG, Düren, Jerman). Akhirnya, plasmid diseleksi terlebih dahulu melalui pencernaan restriksi dengan EcoRI-HF dan HindIII-HF (New England Biolabs Inc., Ipswich, MA, AS) dan diurutkan dengan Sanger (Macrogen). Hasil pengurutan diselaraskan dengan genom referensi lalat buah versi 2.1 (GCF_000347755.3) (Papanicolaou et al. , 2016 ).
Konstruksi gen minimal pupa putih
Konstruksi konstruksi gen minimal untuk gen pupa putih (disebut mini- wp ) melibatkan amplifikasi wilayah 2 kb di hulu 5′ UTR, yang diasumsikan mengandung promotor, bersama dengan bagian dari 5′ UTR itu sendiri (521 bp). Ini dilakukan dengan menggunakan primer P2237 dan P2238 pada DNA genom (gDNA) yang diekstraksi dari betina dewasa EgII yang masih perawan. Urutan pengkodean, bersama dengan bagian dari 5′ UTR dan 3′ UTR (total 2168 bp), diamplifikasi menggunakan 3 µ L cDNA yang diencerkan 1: 5 dari satu pra-pupa strain EgII, yang disiapkan seperti dijelaskan di atas untuk PCR RACE, menggunakan primer P2239 dan P2240 yang membentang dari 5′ UTR yang dianotasi hingga akhir 3′ UTR (Gbr. S1 ). Phusion Flash High-Fidelity PCR Mastermix digunakan untuk amplifikasi dan kondisi siklus sebagai berikut: 98 °C selama 10 detik, 35 siklus 98 °C selama 1 detik, 50 °C selama 5 detik, 72 °C selama 1 menit, 72 °C selama 3 menit. Produk PCR dianalisis melalui elektroforesis gel, diekstraksi menggunakan Zymoclean Gel DNA Recovery Kit (Zymo Research Europe GmbH, Freiburg, Jerman), dan dirakit menjadi vektor transformasi piggyBac yang dicerna SacII/XhoI AH465 ( pXLBacII_IE1hr5-DsRed.T3-SV40 ) (Li & Handler, 2017 ) menggunakan Gibson Assembly Cloning Kit (New England Biolabs Inc., Ipswich, MA, AS). Plasmid M6620 yang dihasilkan ( pXLBacII_mini-wp_IE1hr5-DsRed.T3-SV40 ) digunakan untuk mentransformasi sel XL1-Blue MR Supercompetent (Agilent Technologies, Santa Clara, CA, AS). Perakitan dan urutan sisipan mini- wp yang benar (total 4689 bp, termasuk 2000 bp dari daerah promotor putatif) diverifikasi melalui pencernaan restriksi dengan NgoMIV dan AfiII serta sekuensing Sanger. Akhirnya, DNA plasmid bebas endotoksin disiapkan menggunakan kit pemurnian NucleoBond ® Xtra Maxi EF (Macherey-Nagel GmbH & Co. KG, Düren, Jerman), mengikuti petunjuk pabrik pembuatnya.
Transformasi garis keturunan
Transformasi germline dicapai melalui mikroinjeksi ke dalam embrio strain C. capitata wp −(CRISPR) (Ward et al. , 2021 ), menggunakan campuran buffer KCl (5 mmol/L) dan NaPO4 ( 0,1 mmol/L) pada pH 6,8, plasmid donor piggyBac M6620 (500 ng/ µ L), dan plasmid pembantu piggyBac , phsp-pBac (200 ng/ µ L) (Handler & Harrell II, 1999 ). Mikroinjeksi dilakukan dengan mengikuti prosedur standar yang dijelaskan sebelumnya (Handler et al. , 1998 ; Rong & Golic, 2000 ; Aumann et al. , 2018 ). Secara singkat, embrio strain wp −(CRISPR) dikumpulkan hingga 40 menit, didekorionisasi dalam larutan natrium hipoklorit 1,4% (b/b) selama 3 menit, ditempelkan pada pita perekat dua sisi dan ditutup dengan minyak halokarbon 700 (Sigma Aldrich/Merck KG, Darmstadt, Jerman). Jarum suntik dibuat dari kapiler kaca kuarsa silikon (Science Products for Research in Life Science GmbH, Nomor Produk Q100-70-7.5, Hofheim, Jerman) yang dibuat dalam penarik laser P-2000 (Sutter Instruments, Novato, CA, AS). Peralatan suntik terdiri dari mikromanipulator MN-151 (Narishige, Tokyo, Jepang), FemtoJet 4i (Eppendorf, Hamburg, Jerman), dan mikroskop stereo SZX16 (Olympus, Tokyo, Jepang). Setelah penyuntikan, embrio ditempatkan dalam ruang oksigen dengan kertas saring yang dibasahi pada suhu 21 °C. Larva yang menetas dipindahkan dengan hati-hati ke dalam makanan larva dan dibesarkan dalam kondisi standar pada suhu 25 °C. Lalat yang menetas disilangkan kembali dengan galur induknya, dan keturunannya dikawinkan untuk menghasilkan galur yang diselamatkan.
Penyaringan dan akuisisi gambar
Untuk mengidentifikasi keturunan yang telah ditransformasi, pupa disaring berdasarkan warna WT dari puparia dan lalat, yang dibius dengan CO 2 , disaring berdasarkan ekspresi fluoresensi DsRed di bawah mikroskop stereo Leica M205FC (filter DsRed: eksitasi 530–560 nm, emisi 590–650 nm). Untuk memfasilitasi perolehan gambar, lalat didinginkan di atas es. Gambar diambil menggunakan mikroskop Leica M205 FCA yang digabungkan dengan kamera DMC6200 dan perangkat lunak Leica Application Suite X (versi 3.8.1.26810, Leica, Wetzlar, Jerman).
Karakterisasi molekuler non-mematikan dari penyelamatan wp
Untuk mengonfirmasi secara molekuler fenotipe yang diselamatkan yang diamati, genotipe non-mematikan dilakukan menurut protokol lisis Platinum TM Direct PCR Universal Master Mix (Invitrogen, Thermo Fisher Scientific Baltics UAB, Vilnius, Lithuania). Oleh karena itu, gDNA diekstraksi dari kaki tengah tunggal yang dipotong dengan hati-hati dari lalat dewasa yang dibius dengan CO 2 . Setiap kaki segera dipindahkan ke dalam 20 µ L larutan lisis (mengandung 0,6 µ L proteinase K hingga 20 µ L buffer lisis) dalam tabung mikrocentrifuge dan segera diputar ke bawah untuk memastikan perendaman lengkap dalam larutan. Setiap lalat dipelihara secara terpisah sampai genotipe selesai dan dibiarkan pulih pada kondisi pemeliharaan standar. Supernatan lisis yang mengandung DNA genom digunakan untuk amplifikasi PCR dengan primer P1634 dan P1936, mengikuti petunjuk pabrik pembuatnya. Primer mengamplifikasi bagian dari urutan pengkodean dari ekson pengkodean 1 hingga 3 gen pupa putih , yang mencakup 135 bp intron. Oleh karena itu, ukuran amplikon yang diharapkan untuk gen wp endogen adalah 786 bp, sedangkan amplikon dari mini -wp diharapkan berukuran 651 bp. Produk PCR dianalisis dengan elektroforesis gel agarosa.
PCR digital tetesan
Bahasa Indonesia: Untuk mengonfirmasi jumlah peristiwa integrasi pada generasi pertama lalat yang ditransformasi secara positif dan keturunan heterozigotnya, PCR digital droplet (ddPCR) dilakukan menggunakan generator droplet Sistem Auto-DG dari Bio-Rad, C1000 Touch Thermal Cycler, dan pembaca droplet BIO-RAD QX200, seperti yang dijelaskan sebelumnya (Häcker et al. , 2023 ). Data dianalisis menggunakan Perangkat Lunak QuantaSoft (Bio-Rad, Hercules, CA, AS). Kaset mini- wp berisi DsRed sebagai gen target. Primer dan probe berlabel ganda yang digunakan adalah P49, P50, dan DsRed-probe. Medfly His3 (LOC101459256, yang mengodekan histon H3.3) berfungsi sebagai gen housekeeping referensi. Primer dan probe berlabel ganda meliputi P101, P103, dan CCHis3-probe. Pencernaan restriksi DNA sebelum pembuatan droplet dilakukan menggunakan EcoRI. Setiap campuran reaksi mencakup DNA genomik dari sampel heterozigot, ddPCR 2× Supermix untuk probe (tanpa dUTP, Bio-Rad), campuran primer-probe, dan EcoRI, semuanya mencapai konsentrasi akhir 1× Supermix, oligonukleotida 900 nmol/L, probe 250 nmol/L, dan 2 U EcoRI dalam volume total 25 µ L dalam pelat ddPCR 96-well (Bio-Rad). Kondisi siklus untuk droplet yang dihasilkan adalah sebagai berikut: 95 °C selama 10 menit, diikuti oleh 40 siklus pada 94 °C selama 30 detik dan 55 °C selama 1 menit. Enzim dinonaktifkan pada 98 °C selama 10 menit sebelum fluoresensi droplet dianalisis menggunakan pembaca droplet.
PCR terbalik dan konfirmasi urutan situs integrasi
PCR terbalik (iPCR) dilakukan untuk menentukan lokasi genom insersi piggyBac . 600 ng gDNA, yang diekstraksi dari lalat perawan tunggal, dicerna dengan MspI (New England Biolabs Inc., Ipswich, MA, AS) dalam reaksi 20- µ L pada suhu 37 °C selama 1 jam. DNA yang dicerna diendapkan dalam 3 mol/L NaOAc dan etanol, dan dipulihkan dalam 50 µ L penyangga TE. DNA yang disuspensikan kembali dibiarkan untuk diligasi ulang semalaman (sekitar 18 jam) pada suhu 16 °C dengan 800 Unit DNA ligase T4 (New England Biolabs Inc., Ipswich, MA, AS) dalam reaksi 350- µ L. DNA yang diligasi diendapkan lagi dalam NaOAc dan etanol, dan dipulihkan dalam 50 µ L penyangga TE. Reaksi PCR terbalik dilakukan menggunakan Phusion Flash High-Fidelity PCR Mastermix dan 3 µ L DNA terligasi dalam volume total 20 µ L. Primer mfs11 dan mfs10 digunakan untuk mengamplifikasi daerah yang mengapit situs penyisipan piggyBac 5′, mfs34 dan P815 untuk mengamplifikasi daerah yang mengapit situs penyisipan piggyBac 3′ . Kondisi PCR adalah sebagai berikut: 1× 98 °C 10 dtk; 5× 98 °C 1 dtk, 66−56 °C (5′ piggyBac )/64−54 °C (3′ piggyBac ) (−2 °C per siklus) 5 dtk, 72 °C 60 dtk; 35× 98 °C 1 detik, 56 °C (5′ piggyBac )/54 °C (3′ piggyBac ) 5 detik, 72 °C 60 detik, 1× 72 °C 60 detik. Produk PCR dimurnikan dengan elektroforesis gel dan diekstraksi dengan Zymoclean Gel DNA Recovery Kit (Zymo Research Europe GmbH, Freiburg, Jerman). Akhirnya, produk PCR diurutkan menurut Sanger dan hasilnya dianalisis untuk fragmen vektor piggyBac dan situs restriksi MspI. Urutan tersebut digunakan untuk pencarian BLAST terhadap genom referensi C. capitata (GCF_000347755.3-Genome assembly Ccap_2.1) (Papanicolaou et al. , 2016 ) dan versi assembly EGII-3.2.1 (GCA_905071925.1-Genome assembly EGII-3.2.1) (Ward et al. , 2021 ) menggunakan Geneious Prime. Jika tidak ada atau lemahnya pita yang diamati pada gel agarosa, 1 µ L reaksi iPCR dicadangkan untuk PCR semi-nested (Shen, 2019 ). Dalam kasus ini, produk iPCR diencerkan 1:100 dan 1 µ L digunakan untuk PCR seminested dengan primer mfs10 dan mfs31 untuk piggyBac 5′, atau primer mfs34 dan P139 untuk situs integrasi piggyBac 3′ . Kondisi siklus adalah: 1× 98 °C 10 detik; 35× 98 °C 1 detik, 54 °C (5′ piggyBac )/52 °C (3′ piggyBac ) 5 detik, 72 °C 60 detik, 1× 72 °C 3 menit. Selain itu, situs integrasi dikonfirmasi oleh PCR dengan pengikatan primer di daerah genom (TabelS2 ) mengapit situs integrasi yang dipasangkan dengan primer lain dalam transgen, diikuti oleh sekuensing Sanger dari amplikon yang dihasilkan. Reaksi PCR dilakukan menggunakan Phusion Flash High-Fidelity PCR Mastermix dan 40 ng gDNA dalam reaksi 20- µ L.
Remobilisasi kaset mini- wp piggyBac
Untuk menghasilkan galur dengan situs integrasi genomik baru dari konstruksi mini- wp piggyBac , kami menggunakan galur piggyBac -Jumpstarter 3 (Schetelig et al. , 2009 ) sebagai sumber transposase. Galur ini mengandung kaset pMi{Ccwhite+; hspBac} (AH_370) (Schetelig et al. , 2009 ), yang mengekspresikan transposase piggyBac dan gen medfly white . Pengaturan ini memungkinkan remobilisasi konstruksi piggyBac seperti mini- wp dan penyelamatan fenotipe mata putih. Akibatnya, warna mata dapat digunakan sebagai penanda untuk mengonfirmasi keberadaan kaset Jumpstarter setelah remobilisasi, khususnya saat menggunakan galur dengan fenotipe mata putih dalam percobaan. Oleh karena itu, pertama-tama perlu untuk mendapatkan penyelamatan mini- wp dalam latar belakang mata putih/pupa putih : Galur mini- wp dengan situs integrasi asli disilangkan dengan galur yang homozigot ganda untuk mutasi alami mata putih dan pupa putih ( we − wp − / we − wp − ). Setelah membentuk galur penyelamatan mini- wp dengan latar belakang we − wp − / we − wp − , jantan dan betina galur ini dikawinkan dengan rekan-rekan mereka dari galur Jumpstarter 3. Keturunannya disaring untuk fluoresensi DsRed, warna mata, dan digenotipe secara non-mematikan dengan primer yang mengikat DNA genom yang mengapit situs integrasi asli dan primer di dalam kaset mini- wp untuk menentukan apakah kaset tersebut tetap berada di situs integrasi asli atau mungkin dimobilisasi ulang. Primer P2357, P2358, dan P2273 digunakan pada ujung piggyBac 5′ , sementara mfs34 dan P2359 digunakan pada ujung piggyBac 3′ . Untuk memudahkan identifikasi lalat selama genotipe dan persilangan, semua lalat diberi nomor berurutan dan famili yang terbentuk diberi nama sesuai nomornya. Famili yang terbentuk dari lalat dengan situs integrasi asli diberi nama sesuai nomor plasmid (M6620). Lalat dengan situs integrasi baru yang potensial disilangkan secara individual dengan padanan galur we − wp − / we − wp − . Keturunannya disaring lagi dan lalat yang diselamatkan dengan fenotipe mata putih disilangkan secara individual dengan we− wp − / we − wp − galur. Fenotipe mata putih menunjukkan tidak adanya kaset Jumpstarter, oleh karena itu, menunjukkan bahwa situs integrasi baru telah berhasil distabilkan. Keturunannya kemudian disaring lagi dan lalat yang diselamatkan dikawinkan untuk membentuk galur lalat mini- wp yang diselamatkan di latar belakang we − wp − / we − wp − dengan situs integrasi yang berbeda dari kaset piggyBac (Gbr. 1 ). Situs integrasi galur baru diidentifikasi melalui iPCR dan dibandingkan dengan urutan genom lalat buah yang tersedia, seperti yang dijelaskan di atas. Urutan sisi situs integrasi untuk semua galur mini- wp diberikan dalam Tabel S2 .
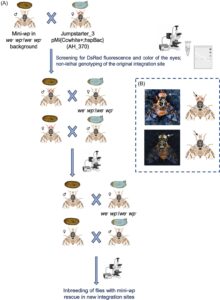
Kemampuan penyelamatan fungsional mini -wp dari posisi genomik yang berbeda
Untuk memverifikasi fungsionalitas konstruksi mini- wp untuk menyelamatkan fenotipe pupa putih ketika diintegrasikan dalam posisi genomik yang berbeda, lalat homozigot yang mengekspresikan penyelamatan mini- wp disilangkan dengan rekan homozigot mereka dari strain yang membawa mutasi alami gen pupa putih . Telur dikumpulkan pada dua hari berturut-turut antara hari oviposisi 5 hingga 8, dalam dua hingga enam kelompok, tergantung pada jumlah total telur yang diletakkan. Idealnya, 100 telur dikumpulkan dari setiap kandang per kelompok. Setelah pengumpulan, setiap kelompok telur dipelihara secara terpisah. Pupa disaring untuk fluoresensi DsRed dan warna puparium dan dihitung sebagai “diselamatkan” (puparium coklat) atau “pupa putih” (puparium putih). Orang dewasa heterozigot yang keluar dari pupa yang diselamatkan disaring untuk fluoresensi DsRed dan sekali lagi disilangkan dengan rekan homozigot mereka dari strain dengan mutasi alami gen wp . Sekali lagi, pupa disaring dan dihitung sebagai pupa yang diselamatkan atau pupa putih. Kemampuan penyelamatan diberikan sebagai persentase pupa dengan fenotipe yang diselamatkan dalam kaitannya dengan jumlah total pupa yang diperoleh. Jumlah telur, pupa, dan dewasa yang dikumpulkan untuk setiap percobaan diberikan dalam Tabel S3–S5 .
Analisis statistik
Analisis data dilakukan menggunakan perangkat lunak MiniTab ® (Minitab, LLC., State College, CA, AS). Data dianalisis melalui analisis varians satu arah (ANOVA), dengan uji Ryan–Joiner untuk kenormalan dan uji Levene untuk varians yang sama. Rata-rata kemudian dibandingkan dengan uji simultan Tukey untuk perbedaan rata-rata pada tingkat kepercayaan 95%. Semua analisis statistik disediakan dalam Tabel S6 .
Hasil
Analisis struktur gen pupa putih dan kloning fragmen gen minimal ke dalam vektor piggyBac
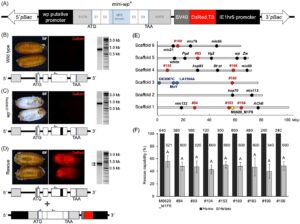
Struktur gen pupa putih (LOC101451947) (Ward et al. , 2021 ), yang dianotasi secara silico dengan panjang total 20868 bp dan urutan pengkodean (CDS) sepanjang 1596 bp pada empat ekson pengkodean, berhasil diverifikasi melalui PCR Amplifikasi Cepat Ujung cDNA (RACE). Pemetaan urutan yang diperoleh ke genom referensi medfly (GCF_000347755.3) (Papanicolaou et al. , 2016 ) menempatkan awal 5′ UTR pada 18489 bp di hulu awal CDS yang diprediksi, ditandai oleh kodon awal, seperti yang disarankan dalam versi mRNA yang dianotasi XM_004530458.4. Di sisi lain, 3′ UTR dipetakan ke versi XM_020860083.1. Karena urutan kargo yang lebih pendek dapat memfasilitasi transgenesis serangga, versi gen minimal dari gen pupa putih (mini -wp , mwp ) dirancang untuk membantu upaya transgenesis serangga berikutnya. Daerah promotor endogen (2000 bp), bersama dengan 5′ UTR, CDS dan 3′ UTR dari gen pupa putih , dengan total 4689 bp, dikloning ke dalam vektor piggyBac yang berisi penanda fluoresensi DsRed (Gbr. 2A ).
Transformasi germline dengan mini- wp mengembalikan fenotipe pupa tipe liar
Transformasi germline dilakukan melalui mikroinjeksi ke dalam 849 embrio strain wp −(CRISPR) (Ward et al. , 2021 ). Semua dewasa G 0 , delapan jantan dan delapan betina, secara individual disilangkan balik dengan strain parental wp −(CRISPR) . Keturunan (G 1 ) disaring untuk fluoresensi DsRed dan warna puparium pada tahap pupa dan satu dari 124 pupa dari betina #6 menunjukkan fenotipe yang diselamatkan, yaitu warna puparium coklat tipe liar dan ekspresi fluoresensi DsRed. Jumlah salinan transgen pada individu yang diselamatkan dan keturunan heterozigotnya dikonfirmasi sebagai salinan tunggal oleh ddPCR, yang menunjukkan peristiwa integrasi piggyBac yang unik . Lalat jantan yang diselamatkan (M6620_M1F6) disilangkan kembali dengan lalat betina dari galur wp −(CRISPR) dan keturunannya dikawinkan untuk membentuk galur yang diselamatkan (Gbr. 2D dan S2 ). Amplifikasi PCR menggunakan primer yang menjangkau dua intron antara ekson pengodean 1 dan 3 dari gen wp , yang dilakukan pada gDNA, memungkinkan untuk membedakan secara molekuler apakah warna puparium cokelat disebabkan oleh rekayasa mini- wp atau alel tipe liar (WT) dari gen wp (Gbr. 2B − D ).
Remobilisasi yang dimediasi transposase mengintegrasikan mini- wp di posisi genom yang berbeda
Untuk memverifikasi bahwa konstruksi penyelamatan mini- wp dapat diintegrasikan dan berfungsi dalam posisi genomik lain dalam genom medfly, galur Jumpstarter 3 (Schetelig et al. , 2009 ) digunakan sebagai sumber transposase piggyBac untuk memobilisasi kembali kaset mini -wp dari situs integrasi aslinya. Mengikuti skema persilangan yang digambarkan dalam Gambar 1 , delapan galur baru dengan fenotipe yang diselamatkan telah terbentuk. Situs integrasi untuk semua galur yang diselamatkan ditentukan melalui iPCR, dan wilayah genom yang mengapit situs integrasi dikonfirmasi oleh sekuensing Sanger (Tabel S2 ). Khususnya, lokasi integrasi strain #160 diidentifikasi dalam wilayah perancah yang diprediksi berada pada kromosom X. Lokasi integrasi semua strain lainnya diidentifikasi dalam posisi autosomal, dan berdasarkan analisis in silico dari gen yang diberi anotasi dan urutan mikrosatelit (Papanicolaou et al. , 2016 ), kemungkinan berada pada kromosom 2 (perancah 1), 4 (perancah 6), 5 (perancah 5), dan 6 (perancah 4) (Gbr. 2E ).
Mini- wp berhasil menyelamatkan fenotipe pupa WT dari situs integrasi yang berbeda
Bahasa Indonesia: Untuk mengevaluasi secara fungsional penyelamatan mini- wp yang terintegrasi dalam posisi genomik yang berbeda, lalat dari galur yang diselamatkan disilangkan dengan lalat yang membawa mutasi alami gen pupa putih , yang dilaporkan sebagai penyisipan ∼8150 bp pada ekson pengodean ketiga (Ward et al. , 2021 ). Keturunannya disaring untuk fenotipe WT yang diselamatkan dan fluoresensi DsRed pada tahap pupa dan dewasa disilangkan lagi untuk mengevaluasi kemampuan penyelamatan dalam heterozigositas. Penyelamatan dari semua posisi yang dievaluasi berfungsi penuh, dengan 100% keturunan dari persilangan homozigot menunjukkan fenotipe WT yang dipulihkan pada tahap pupa. Selain itu, penyelamatan juga berfungsi dalam heterozigositas, yaitu, satu salinan gen mini- wp cukup untuk menyelamatkan mutan wp – homozigot (Gbr. S3 ). Konsisten dengan pola pewarisan Mendel, ∼50% dari keturunan mengekspresikan fenotipe pupa yang diselamatkan ketika menyilangkan lalat m wp ±(nat) dengan mutan wp −(nat)/-(nat) (47,43% ± 4,5%, rata-rata di semua galur dengan situs integrasi yang berbeda dari mini- wp , tanpa perbedaan statistik yang signifikan antara galur ( nilai- P = 0,297, ANOVA satu arah). Untuk memvalidasi secara fungsional integrasi yang diprediksi berada pada kromosom X, kami menyilangkan jantan dari galur #160 dengan rekan-rekan mereka yang membawa mutasi alami pada gen wp . Seperti yang diharapkan untuk penanda yang terletak pada kromosom X, semua keturunan yang diselamatkan adalah betina, sementara semua lalat yang keluar dari puparia putih adalah jantan (Tabel 1 ). Menariknya, dalam putaran pemeliharaan berikutnya untuk pemeliharaan garis #160, ketika menyilangkan keturunan jantan yang diselamatkan dengan betina dari galur wp – , kami mengamati dua jantan yang menetas dari pupa yang diselamatkan. Dalam kesempatan ini, total 600 telur dikumpulkan. Berdasarkan hipotesis bahwa peristiwa rekombinasi mungkin telah terjadi, jantan ini disilangkan dengan betina dari galur wp − . Meskipun beberapa telur dikumpulkan, tidak ada keturunan dewasa yang diperoleh dan tidak mungkin untuk membentuk keluarga baru dari lalat jantan ini. Karena jantan ini tidak bertahan hidup atau menghasilkan keturunan, tidak ada analisis lebih lanjut yang dilakukan sebagai bagian dari pekerjaan ini. Galur #104 dan #183 tidak dapat bertahan hidup setelah sekitar lima generasi perkawinan sedarah, yang menunjukkan bahwa integrasi kaset penyelamat di galur yang berbeda mungkin memiliki biaya kebugaran, yang memengaruhi pemeliharaannya.
| Fenotipe | Jumlah pupa | Jumlah jantan yang menetas | Jumlah betina yang menetas |
|---|---|---|---|
| Diselamatkan | 208 | angka 0 | 200 |
| Kepompong putih | 263 | 247 | angka 0 |
Fenotipe tak terduga yang diperoleh dari remobilisasi integrasi piggyBac
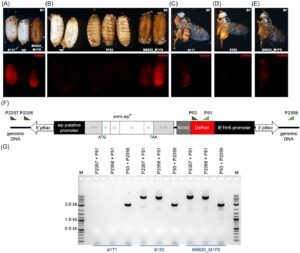
Di antara lalat yang diperoleh dari percobaan remobilisasi, lalat #171 dan #193 menonjol dengan fenotipe yang tidak terduga. Lalat #171 tidak menunjukkan fenotipe warna puparium yang diselamatkan, tetapi ekspresi positif dari penanda fluoresensi (Gbr. 3A, C ). Amplifikasi PCR yang mengapit posisi genomik dari situs integrasi asli menunjukkan amplifikasi parsial dari kaset pada ujung 3′ dari kaset terintegrasi pada posisi asli. Namun, amplifikasi yang mencakup ujung 5′ dari kaset tidak memungkinkan, yang menunjukkan bahwa bagian dari konstruksi tersebut hilang (Gbr. 3G ). Itu juga tidak memungkinkan untuk memperkuat bagian dari kaset mini- wp di hulu dari penanda fluoresensi (Gbr. S4 ), yang menunjukkan bahwa bagian dari kaset mungkin telah dikeluarkan. Lalat #193 menunjukkan fenotipe WT tersegmentasi dari puparium (Gbr. 3B ), dan tidak ada ekspresi penanda fluoresensi (Gbr. 3B , D ). Amplifikasi PCR pada ujung 3′ dan 5′ dari kaset penyelamat mengonfirmasi situs integrasi strain asli (M6620_M1F6), yang menunjukkan bahwa tidak ada remobilisasi (Gbr. 3G ).
Diskusi
Konsep GSS neo-klasik menyediakan pendekatan generik untuk merekayasa GSS di berbagai spesies hama serangga dengan cara yang terarah dan hemat waktu (Häcker et al. , 2021 ; Nguyen et al. , 2021 ; Yan et al. , 2023 ; Yan et al. , 2024 ). Tidak seperti GSS klasik, yang bergantung pada mutasi acak atau yang diinduksi, pendekatan neo-klasik menggunakan penyuntingan genom untuk presisi dan efisiensi yang lebih tinggi (Ward et al. , 2021 ; Chen et al. , 2022 ; Sollazzo et al. , 2024 ). CRISPR/Cas dapat menginduksi mutasi yang ditargetkan pada gen penanda, memfasilitasi galur mutan fenotipik. Selain itu, pendekatan ini dapat mengatasi masalah semisterilitas dan rekombinasi yang terkait dengan translokasi yang disebabkan oleh iradiasi dengan mengintegrasikan secara tepat alel WT ke dalam kromosom Y atau di dekat faktor penentu laki-laki (Franz et al. , 2021 ; Cáceres et al. , 2023 ).
Di sini, kami menyelidiki gen pupa putih sebagai penanda potensial untuk pendekatan generik guna membangun GSS neo-klasik dan menunjukkan bahwa fenotipe warna pupa coklat tipe liar dapat dipulihkan melalui penyisipan versi nir intron dari gen wp (mini- wp) yang dimediasi piggyBac ke dalam genom strain mutan pupa putih .
Penelitian sebelumnya menunjukkan bahwa efisiensi knock-in yang dimediasi HDR menurun seiring dengan peningkatan ukuran kargo (Li et al. , 2014 ; Paix et al. , 2017 ), dan bahwa insersi yang ditargetkan mungkin sangat menantang di daerah berulang dan heterokromatik, seperti kromosom Y (Bernardini et al. , 2014 ; Buchman & Akbari, 2019 ). Akibatnya, menggunakan gen pupa putih lengkap untuk menetapkan hubungan seks dalam GSS neo-klasik mungkin menimbulkan tantangan, karena kargo akan mencapai sekitar 20 kb karena intron 18 kb yang terletak di antara 5′ UTR dan kodon awal gen wp . Untuk mengatasi tantangan ini, kami merancang versi minimal tanpa intron dari gen wp dengan hanya 4689 bp, termasuk urutan genom 2000 bp di hulu 5′ UTR yang kami gunakan sebagai wilayah promotor putatif. Integrasi mini- wp yang berhasil dimediasi piggyBac dan pemulihan fenotipe pupa WT yang dihasilkan dalam galur wp −(CRISPR) menunjukkan kelayakan untuk memperoleh penyelamatan fenotipe lengkap menggunakan gen wp tanpa intron . Meskipun tidak diselidiki dalam penelitian ini, mungkin saja untuk mengurangi lebih lanjut ukuran mini-gen dengan memadatkan CDS agar hanya mencakup domain fungsionalnya atau dengan menggunakan daerah promotor endogen yang lebih pendek. Kemungkinan terakhir didukung oleh temuan terkini di mana versi minimal gen wp Z. cucurbitae tetap fungsional setelah daerah promotor dikurangi dari 2000 bp menjadi 605 bp (Fan et al. , 2025 ).
Uji PCR menggunakan primer yang menjangkau intron gen wp memungkinkan diferensiasi antara lalat fenotipe pupa coklat yang membawa alel WT endogen ( wp⁺/wp⁺ ) dan lalat dengan setidaknya satu salinan alel mini- wp yang direkayasa dalam latar belakang mutan pupa putih ( wp ⁻/m wp + ) (Gbr. 2B , D ). Pendekatan ini berharga untuk pengendalian mutu di fasilitas pemeliharaan massal dan untuk mengidentifikasi lalat yang ditangkap kembali dalam program SIT. Lebih jauh lagi, pendekatan ini menghilangkan kebutuhan untuk penanda fluoresensi terkait, yang memungkinkan konstruksi penyelamatan sepenuhnya terdiri dari sekuens endogen, yang dapat memfasilitasi persetujuan regulasi. Jika kerangka regulasi mengecualikan modifikasi yang ditargetkan yang hanya memperkenalkan sekuens yang terjadi secara alami—bahkan dengan perubahan kecil seperti penghapusan kecil—dari klasifikasi GMO, maka serangga yang direkayasa dengan modifikasi minimal tersebut dapat dianggap setara dengan serangga yang dibiakkan secara konvensional dan dengan demikian tidak tunduk pada regulasi GMO. Hal ini dapat memiliki implikasi yang signifikan terhadap penerimaan regulasi dan persepsi publik.
Selama pemeliharaan strain yang dihasilkan dalam percobaan remobilisasi, kami mengamati variasi dalam kinerja keseluruhan (Tabel S3 ), dengan dua dari sembilan strain (#104 dan #183) tidak dapat dipertahankan. Variabilitas ini kemungkinan berasal dari sifat acak integrasi piggyBac , karena penyisipan ke gen esensial atau daerah pengatur dapat mengganggu ekspresi gen dan merusak kebugaran. Pada strain #104, misalnya, situs integrasi m wp terletak di dalam gen pengkode protein yang diprediksi, protein 2 yang mengandung domain pengikat nukleotida siklik, yang mungkin telah memengaruhi viabilitas. Meskipun demikian, tujuh strain lainnya telah dipertahankan dalam kondisi laboratorium dan secara stabil mengekspresikan fenotipe WT yang dipulihkan selama lebih dari sepuluh generasi. Pada strain #171, fenotipe pupa putih disertai dengan ekspresi DsRed (Gbr. 3A , G ) menunjukkan eksisi parsial kaset m wp , hanya menyisakan penanda fluoresensi dan ujung 3′ vektor piggyBac di situs asli. Demikian pula, fenotipe bergaris WT yang diamati pada galur #193 mungkin disebabkan oleh mutasi yang memengaruhi wilayah regulasi wp atau elemen genetik lainnya. Namun, mutasi ini tidak menjelaskan tidak adanya ekspresi penanda fluoresensi. Hubungan antara fenotipe ini dan percobaan remobilisasi masih belum jelas, dan penyelidikan lebih lanjut berada di luar cakupan penelitian ini.
Untuk memajukan rekayasa GSS neo-klasik, teknologi CRISPR/Cas dapat digunakan untuk mengintegrasikan mini -wp secara tepat ke dalam wilayah kromosom Y tertentu pada galur fenotip pupa putih , memastikan ekspresi yang stabil dan spesifik jenis kelamin. Mencapai sekuensing kromosom Y berkualitas tinggi dan menetapkan protokol yang andal untuk penyuntingan gen Y yang tepat adalah tantangan berikutnya, bersama dengan mengidentifikasi lokasi target optimal pada kromosom Y untuk memastikan ekspresi yang kuat dan integrasi konstruksi yang stabil. Fondasi yang kuat dari fungsi gen dan penelitian penyuntingan gen dalam C. capitata (Meccariello et al. , 2017 ; Aumann et al. , 2018 ; Meccariello et al. , 2019 ; Aumann et al. , 2020 ; Meccariello et al. , 2021 ; Ward et al. , 2021 ; Sollazzo et al. , 2024 ) menggarisbawahi pentingnya spesies ini untuk eksperimen pembuktian konsep dan untuk pengembangan lebih lanjut GSS neo-klasik. Selain itu, GSS klasik yang mapan, VIENNA-7 dan VIENNA-8, dikombinasikan dengan pengalaman luas dalam pemeliharaan massal, kemungkinan akan memfasilitasi eksperimen skala besar di masa mendatang, termasuk penilaian kebugaran pada strain rekayasa ini. Perangkat yang dikembangkan untuk merancang dan memvalidasi gen wp tanpa intron dapat diperluas ke spesies terkait tempat gen ini telah diidentifikasi dan galur wp ⁻(CRISPR) telah dihasilkan (Ward et al. , 2021 ; Paulo et al. , 2022 ). Memperluas pendekatan ini ke berbagai spesies target SIT akan meningkatkan fleksibilitas dan penerapan GSS neoklasik.
Secara keseluruhan, temuan kami merupakan langkah signifikan menuju pengembangan GSS neo-klasik untuk aplikasi SIT. Kami telah menunjukkan bahwa fenotipe WT dapat dipulihkan dengan mengintegrasikan gen wp tanpa intron ke berbagai situs genomik di Ceratitis capitata . Studi ini memberikan dasar bagi upaya penyuntingan gen di masa mendatang untuk merekayasa GSS pada spesies target SIT lainnya, memanfaatkan perangkat molekuler modern untuk mengembangkan penanda yang dapat dipilih berdasarkan jenis kelamin yang kuat dan efisien. Penyempurnaan lebih lanjut, termasuk mengoptimalkan situs integrasi dan menilai stabilitas jangka panjang, akan sangat penting untuk memajukan pendekatan ini menuju implementasi praktis.



Tinggalkan Balasan